|
 |
| Res. Plant Dis > Volume 27(3); 2021 > Article |
|
요 약
식물 바이러스는 작물 수확량에 상당한 손실을 일으키고 작물 생산을 지속적으로 위협하여 세계 식량 안보에 심각한 위협이 된다. 그 중 tomato spotted wilt virus (TSWV)는 주로 원예 작물을 감염시키는 가장 위협적인 식물 바이러스로 넓은 기주 범위를 가진다. Reverse-transcription quantitative real-timePCR (RT-qPCR)은 TSWV의 민감한 검출을 위해 널리 사용되고 있지만 표준화의 어려움으로 인해 유용성이 감소한다. 따라서 본 연구에서는 TSWV 검출을 위해 민감하고 정확한 reverse transcription droplet digital polymerase chain reaction (RT-ddPCR)을 확립하였다. TSWV 검출에 대한 RT-qPCR 및 RT-ddPCR의 민감도를 비교하였고, TSWV에 대한 RT-ddPCR의 특이성 분석은 고추에서 주로 발생하는 바이러스 및 음성 대조군에서 특이성을 확인한 결과 증폭되지 않았다. RT-ddPCR 및 RTqPCR에 의해 측정된 TSWV의 선형회귀곡선은 모두 높은 선형성을 나타냈지만, RT-ddPCR 분석이 10배 이상 더 민감하고 더 낮은 TSWV의 copy 수를 검출할 수 있었다. 종합적으로, 우리의 연구 결과는 RT-ddPCR이 TSWV 검출에 대해 높은 민감도와 특이성을 제공하고 낮은 농도의 현장 시료에서 TSWV 검출하는데 적합하다는 것을 보여준다.
ABSTRACT
Plant viruses cause significant yield losses, continuously compromising crop production and thus representing a serious threat to global food security. Tomato spotted wilt virus (TSWV) is the most harmful plant virus that mainly infects horticultural crops and has a wide host range. Reverse-transcription quantitative real-time PCR (RT-qPCR) has been widely used for detecting TSWV with high sensitivity, but its application is limited owing to the lack of standardization. Therefore, in this study, a sensitive and accurate reverse transcription droplet digital polymerase chain reaction (RT-ddPCR) method was established for TSWV detection. Additionally, we compared the sensitivities of RT-qPCR and RT-ddPCR for TSWV detection. Specificity analysis of RT-ddPCR for TSWV showed no amplification for main pepper viruses and negative control. TSWV transcripts levels measured by RT-ddPCR and RT-qPCR showed a high degree of linearity; however, the former yielded results that were at least 10-fold more sensitive and detected lower TSWV copy numbers than the latter. Collectively, our findings show that RT-ddPCR provides improved analytical sensitivity and specificity for TSWV detection, making it suitable for identifying low TSWV concentrations in field samples.
식물 바이러스병은 전 세계적으로 다양한 작물에서 양적 및 질적으로 큰 피해를 야기하고 있다. 비록 전 세계적으로 바이러스 병에 의한 경제적 손실을 정확히 계산하기는 어렵지만, 매년 발생하는 경제적 손실액은 32조 550억원으로 추정된다(Sastry and Zitter, 2014). 다년생 작물과 과수의 바이러스 피해는 작물의 생산량을 감소시킬 뿐만 아니라 재배 시간 및 비용 손해도 고려되어야 할 것이다(Hull, 2013). 종종 단독 바이러스 감염에
서는 병징이 보이지 않아 바이러스 병 피해가 크게 없을 것이라 과소평가되고 있는 바이러스도 있지만 다른 병원균들과 복합감염이 되면서 큰 경제적 손실을 야기시키는 경우도 있다(Hull, 2013). 따라서 식물바이러스는 전 세계적으로 작물 피해, 국제 식량문제 등 인간 삶에 있어 전반적으로 위협요인으로 예방 및 방제 전략이 필요하다.
고추에 발생하는 토마토 반점 위조 바이러스(tomato spotted wilt virus, TSWV)는 2003년 예산의 파프리카 재배농가에서 처음으로 발생했다(Kim 등, 2004). 이후 2004년 경기도 안양지역에서 토마토 등의 작물에서 대발생한 후 점차적으로 발생 피해가 증가하고 있는 병원성이 강한 바이러스로서 주로 노지 고추 재배지에서 발생하는데 기주 범위가 넓어 고추 뿐만 아니라 토마토와 파프리카 등 총 1,090종의 식물에서 보고되고 있다(Cho 등, 2005; Parrella 등, 2003). TSWV에 감염된 식물은 잎에 괴사증상이 나타나고 과실의 크기가 작아지며 기형이 되고 색이 얼룩덜룩하여 고르게 착색이 되지 않기 때문에 고추 칼라병이라고도 불리며 상품성이 전혀 없다(Kurstak, 1981; Oetting, 1991; Peters 등, 1991). 현재 국내에서는 TSWV 피해예방을 위한 농약 살포와 저항성 품종 개발에 의존하고 있는 상황이다(Han 등, 2011, 2012). 최근 기상변화와 재배법의 변화, 종자 생산 기술, 농산물의 국제교류 확대에 따른 국내에서 발생하지 않았던 고위험 바이러스의 국내유입 및 피해확산 사례가 지속적으로 발생함에 따라 경제적 피해도 매우 큰 상황이기 때문에 정확하고 민감한 대응기술 마련이 시급하다.
현재까지 TSWV를 검출하기 위해서는 효소면역측정법, 역전사 중합효소 연쇄반응(reverse transcription polymerase chain reaction, RT-PCR), 역전사 실시간 중합효소 연쇄반응(reversetranscription quantitative real-time polymerase chain reaction, RT-qPCR), 역전사 루프 매개 등온증폭법(reverse transcription-loop-mediated isothermal amplification) 및 스트립 기반 역전사 재조합효소-중합효소 증폭법(reverse-transcription recombinase polymerase amplification assay combined with lateral flow strip) 등이 개발되었다(Cho 등, 1988; Debreczeni 등, 2011; Fukuta 등, 2004; Karavina and Gubba, 2017; Lee 등, 2021b; Roberts 등, 2000). 다양한 기법들 중에서 TSWV를 진단하기 위한 가장 보편적인 방법은 1세대 PCR인 전기 영동 기반 일반 PCR 기법인데, 일반 PCR의 경우 표적 유전자의 copy수까지는 확인할 수 없고, 극소량의 바이러스를 정밀하게 검출할 수 없기 때문에 이를 보완하기 위한 2세대 PCR인 quantitative PCR (qPCR)이 개발되었다(Ali 등, 2012; Floren 등, 2015). 그러나 정량적 분석이 가능한 qPCR은 정량 분석을 위해서는 반드시 농도를 알고 있는 표적 유전자를 통해 얻은 표준곡선이 필요하다는 본질적인 제약이 있다(Debreczeni 등, 2011; Higuchi 등, 1993; Hindson 등, 2011). 따라서 기존의 PCR 기법들의 한계를 극복하기 위해서 3세대 PCR인 디지털 PCR (digital PCR, dPCR)이 개발되었다.
dPCR은 표준 곡선의 사용에 의존하지 않고 표적 서열의 절대 정량수치를 제공하는 새로운 핵산 검출 방법이다(Hindson 등, 2011; Pinheiro 등, 2012; Vogelstein and Kinzler, 1999). 현재 보편적으로 사용되고 있는 dPCR 시스템은 두 가지 접근 방식이 있다(Baker, 2012; Pinheiro 등, 2012). 첫 번째 방식은 시료를 microwells 또는 microfluidic chambers를 통해 수백개로 분할하는 방법이 있고 두 번째 방식은 droplet digital PCR (ddPCR)방식으로 PCR 혼합물을 water-oil emulsion에서 수천 또는 수백만개의 초미세 액체 방울(droplet)로 분할하는 방법이다(Fan and Quake, 2007; Morrison 등, 2006; Ottesen 등, 2006; Warren 등, 2006).
여기서 ddPCR 방법은 PCR 반응을 2만개의 droplet으로 분리하여 증폭시킨 후, 표적 유전자를 계수하는 시스템이다(Fig. 1). Droplet에서의 표적 유전자 증폭 여부에 따라 positive droplet을 1, negative droplet을 0으로 계수하여 푸아송 분포(Poisson distribution)를 통해 표적 유전자의 복제 수를 계산해 최종적으로 시료의 μl당 copy 수를 확인할 수 있다(Dube 등, 2008; Hindson 등, 2011; Nakano 등, 2003; Pinheiro 등, 2012; Yang 등, 2014). 따라서 ddPCR은 기존의 PCR법의 단점을 보완하여 높은 민감도, 신뢰성, 특이성 및 낮은 바이러스 역가를 절대 정량 할수 있기 때문에 분자 생물학 및 역학 연구, 진단 및 방제에 유용한 도구로 사용되고 있는 새로운 접근 방식이다.
최근 많은 연구에서 역전사(reverse transcription, RT) ddPCR 을 이용한 식물 RNA 바이러스의 성공적인 검출 및 정량이 보고되고 있다(Bahder 등, 2016; Gutiérrez-Aguirre 등, 2015; Lee 등, 2021a; Liu 등, 2019; Mehle 등, 2018; Pandey 등, 2020). 따라서 식물 바이러스병을 예방하고 과수, 화훼 및 약용작물의 낮은 농도의 바이러스를 검출할 수 있는 고감도 ddPCR을 이용한 TSWV 진단시스템을 개발하였다.
시료 준비. TSWV 검출에서 사용된 고추 잎은 국내 광주 재배농가에서 채집하였다. 또한 특이성 분석에서 사용된 오이모자이크바이러스(cucumber mosaic virus, CMV), 고추모틀바이러스(pepper mottle virus, PepMoV), 고추약한모틀바이러스(pepper mild mottle virus, PMMoV) 및 잠두위조바이러스2(broad bean wilt virus 2, BBWV2)에 감염된 시료는 국내 광주 재배 농가에서 채집하였다.
핵산 추출. TSWV 감염된 고추 잎에서 RNA를 추출하기 위해 제조사 지침에 따라 Clear-S Total RNA Extraction Kit (Invirustech, Gwangju, Korea)를 사용하여 total RNA를 추출한 후 -80°C의 초저온 냉동고에 보관하였다. 추출한 RNA의 농도는 BioDrop 나노 분광광도계(Biochrom Ltd., Cambridge, UK)를 사용하여 측정하였고, RNA 품질은 RedSafe 핵산 염색이 포함된 1.2% (w/v) agarose gel (iNtRON, Daejeon, Korea)을 사용하여 전기영동을 통해 확인하였다.
시험관 내 전사. TSWV의 외피단백질을 특이적으로 증폭하는 프라이머 세트(Lee 등, 2021b)를 사용하여 RT-PCR로 증폭하였고, 증폭 산물은 AccuPrep PCR/Gel DNA Purification Kit(Bioneer, Daejeon, Korea)를 사용하여 정제하였다. 정제된 PCR증폭산물은 AccuRapid TA Cloning kit를 사용하여 클로닝되었다. 시험관 내 전사는 T7 RNA polymerase (Promega, Madison, WI, USA)를 사용하여 제조사 지침에 따라 수행하였다.
RT 반응. RT-qPCR 및 RT-ddPCR 분석을 위한 RT 반응은 5 μg의 total RNA, 1 μl의 10 mM dNTP, 4 μl의 5× first-strand buffer(250 mM Tris-HCl [pH 8.3], 375 mM KCl, 15 mM MgCl2), 2 μl의 100 mM DTT, 1 μl의 Recombinant RNase Inhibitor (TaKaRa, Shiga, Japan), 1 μl의 100 μM random hexamer primer 및 1 μl의 SuperScript II Reverse Transcriptase (Invitrogen, Carlsbad, CA, USA)를 포함하여 최종 20 μl 부피로 42°C에서 50분, 70°C에서 15분 동안 RT 반응을 수행한 후, AccuPrep PCR/Gel DNA Purification Kit (Bioneer)를 사용하여 합성된 cDNA를 정제하였다. 분석의 일관성을 보장하기 위해 RT-qPCR 및 RT-ddPCR 분석 모두에 합성된 cDNA를 사용하였다.
프라이머 및 TaqMan 프로브 제작. BioEdit 소프트웨어를 사용하여 777 bp의 TSWV 외피단백질 염기서열을 기반으로 GenBank 데이터베이스에서 얻은 17개의 국내외 TSWV 분리주(MK605252, MF156073, MG254896, MN064724, MF159066, KY021439, MF159068, LC273307, MF417648, MF159062, KU179571, KU17941, KU17937, MG983521, MG983519, MG878857, MF688996)를 정렬한 후 보존된 영역에서 RT-qPCR과 RT-ddPCR용 프라이머 및 프로브를 설계하였다. 설계된 프라이머 및 프로브 염기서열은 정방향 프라이머 5'-GCAAGCCTCACAGACTTTGC-3', 역방향 프라이머 5'-TGGAGCCACTGACATGACCT-3' 및 프로브 6-FAM-CCCAGTTTCCTCRACAAGYCTGACCC-BHQ-1이며 모두 Bionics (Daejeon, Korea)에서 합성되었다.
TaqMan 기반 RT-qPCR 분석. RT-qPCR 혼합물은 10 μl의 qPCR 2× Master mix (TaqMan Probe, ELPIS-Biotech, Daejeon, Korea), 1 μl의 정방향 및 역방향 프라이머(10 pmol), 0.5 μ l의 프로브(10 pmol) 및 1 μl의 TSWV-cDNA를 포함하여 최종부피 20 μl로 BioRad CFX96 Real-Time PCR (Bio-Rad, Hercules, CA, USA)를 사용하여 분석하였다. RT-qPCR 반응은 동일한 조건에서 3반복으로 분석되었다. qPCR 조건은 95°C에서 15분간 초기 변성 후 95°C에서 20초, 60°C에서 40초, 72°C에서 30초를 3단계로 40회 반복하였다.
RT-ddPCR 분석. RT-ddPCR은 제조사의 지침에 따라 QX200 Droplet Digital PCR system (Bio-Rad)을 사용하여 수행하였다. RT-ddPCR 혼합물은 10 μl의 2× ddPCR Supermix for Probes (Bio-Rad), 900 nM의 정방향 및 역방향 프라이머, 250 nM의 프로브, 4.9 μl의 DEPC-water 및 1 μl의 TSWV-cDNA를 포함하여 최종 부피 20 μl로 분석되었다. 오염을 확인하기 위해 음성 대조군으로 TSWV가 없는 negative control (NTC)과 함께 적어도 3반복으로 분석되었다. 각 시료에 대한 20 μl의 PCR 혼합물과 70 μl의 Droplet Generation Oil for Probes (Bio-Rad)를 DG8 cartridge (Bio-Rad)에 분주하고, DG8 Gaskets (Bio-Rad)로 덮어 QX200 droplet generator (Bio-Rad)에서 각 well의 droplet을 생성하였다. 각 well의 생성된 40 μl의 droplet는 8-Channel VIAFLO Electronic Pipette (INTEGRA, Hudson, NH, USA)를 사용하여 96-well PCR plate (Bio-Rad)로 옮겼다. PCR plate는 PX1 PCR plate sealer (Bio-Rad)를 사용하여 Pierceable Foil Heat Seal (Bio-Rad)로 밀봉한 다음 T100 Thermal Cyclerv (Bio-Rad)에서 증폭하였다. RT-ddPCR 증폭은 95°C에서 10분 동안 초기 변성 단계를 수행한 후 94°C에서 30초 동안 변성, 56°C에서 60초 동안 결합 및 합성(temperature ramp 2°C/sec)를 2단계로 40회 반복하였고 98°C에서 10분, 최종적으로 4°C에서 30분간 보관 후 분석되었다. RT-ddPCR에 대한 최적온도를 확인하기 위해 결합 및 합성 단계의 온도를 48-62°C로 설정하였다. 각 시료의 copy 수는 설정된 임계값(threshold)에 따라 양성 및 음성 형광 신호를 분리한 후 QuantaSoft software version 1.7.4 (Bio-Rad)에 의해 분석되었다. RT-ddPCR 결과는 15,000개 이상의 droplet이 포함된 결과만 사용되었다. 각 well 에 대해 계산된 오차 막대는 95%의 신뢰 구간을 나타낸다.
RT-ddPCR 특이성 평가. RT-ddPCR 분석법의 특이성과 교차 반응성은 국내 고추에서 발생하는 CMV, PepMoV, PMMoV, BBWV2 감염 시료와 음성대조군인 고추 건전주와 NTC를 사용하여 평가하였다. 분석 내 변동성을 평가하기 위해 각 실험은 3회 반복되었고, 분석 간 변동성을 평가하기 위해 3번의 독립적인 분석을 수행하였다.
민감도 비교 및 통계 분석. RT-qPCR 및 RT-ddPCR 분석의 민감도를 비교하기 위해 TSWV-cDNA를 10배씩 연속 희석한 시료를 사용하여 3번의 독립적인 실험을 3회 반복하여 분석하였다. RT-qPCR과 RT-ddPCR 분석의 변동 계수(coefficient of variation, CV)는 3회 반복의 표준 편차/평균으로 계산되었다. 선형 회귀분석은 시료의 Cq값과 concentration으로부터 각각 분석되었고, 선형 결정 계수(R2)도 RT-ddPCR 및 RT-qPCR에 대해 계산되었다.
RT-ddPCR을 위한 annealing 온도 최적화. RT-ddPCR에 대한 최적의 annealing 온도를 결정하기 위해 동일한 양의 TSWV-cDNA를 사용하여 48°C에서 62°C까지의 각각 다른 온도에서 RT-ddPCR 분석을 수행하였다. 음성 droplet과 양성 droplet의 분리된 형광 진폭을 확인하여 가장 적합한 온도 조건을 확인하였고 56.5°C를 최적온도를 결정하였다(Fig. 2). 48°C에서 53.4°C의 낮은 annealing 온도는 형광 진폭의 차이로 인한 분리정도가 높았지만 양성 droplet과 음성 droplet 사이의 특이성이 감소하였고, 반면에 59.1°C부터 62°C의 높은 annealing 온도는 특이성은 높았지만 형광 진폭의 차이가 크지 않았다. 따라서 이후 진행된 실험에서는 56.5°C의 최적 annealing 온도를 사용하여 수행되었다.
RT-ddPCR 분석의 특이성. 국내 고추에서 많이 발생하는 CMV, PepMoV, PMMoV, BBWV2에 감염된 시료를 사용하여 TSWV에 대한 RT-ddPCR 분석의 특이성과 교차 반응성을 평가했으며, 고추 건전주와 NTC는 음성 대조군으로 사용하였다. 특이성 분석 결과 TSWV-cDNA를 사용한 양성 대조군에서는 양성 droplet과 음성 droplet이 명확하게 분리되어 증폭된 반면에 다른 고추 바이러스들과 음성 대조군에서는 양성 droplet은 증폭되지 않고 음성 droplet만 확인되는 것으로 TSWV 검출을 위한 RT-ddPCR 분석이 다른 바이러스들과 교차 반응 없이 TSWV만 특이적으로 증폭됨으로 우수한 특이성을 갖는 것을 확인하였다(Fig. 3).
민감도 분석 비교. RT-qPCR과 RT-ddPCR 분석 간의 민감도를 평가하기 위해 TSWV-cDNA를 10배 연속 희석한 시료를 통해 3회 반복하여 분석하였다. 두 분석의 민감도를 비교한 결과 RT-qPCR의 경우 10-9으로 희석된 시료에서 37.99±0.49의 Cq값까지 검출된 반면에 RT-ddPCR 분석은 10-10으로 희석된 시료에서 2±0.69의 concentration (copies/μl)까지 검출되었다. 따라서 RT-ddPCR 분석의 민감도가 RT-qPCR 분석보다 약 10배 이상 높음을 확인하였고, RT-ddPCR 분석은 TSWV의 1.6 copies/μl의 농도까지 검출이 가능한 것을 판단되었다(Table 1). 또한, RT-qPCR과 RT-ddPCR 분석을 사용하여 평가된 시료에 대해 얻은 CV값의 범위는 각각 0.42-1.3%와 5.4-38.12%로 전체 변동성을 확인하였다(Table 1). RT-ddPCR에 비해 RT-qPCR이 낮은 CV값을 가지는 이유는 CV값은 표준 편차를 평균으로 나눈 값인데, RT-qPCR은 Cq값을 기준으로 계산하였고 RT-ddPCR은 concentration (copies/μl)를 기준으로 계산하였기 때문에 비교적 작은 수의 범위를 가지는 RT-qPCR의 CV값이 더 낮음을 알 수 있다. 또한 RT-qPCR과 RT-ddPCR을 통해 얻은 선형 회귀 곡선을 기반으로 각각 0.9998 및 0.9935의 상관계수 값을 얻었으며, 이는 RT-ddPCR 및 RT-qPCR 분석을 사용하여 얻은 결과가 정량화 범위에서 높은 수준의 선형성 나타내는 것을 알 수 있었다(Fig. 4).
포장 시료에서의 RT-ddPCR 적용. RT-ddPCR의 검출 능력은 RT-qPCR과 비교하여 현장에서 채집된 국내 고추 시료 45개에서 평가되었으며, 검출된 시료를 positive, 검출 안된 시료를 negative로 분류하여 RT-qPCR과 RT-ddPCR에서 검출된 시료의 개수를 동시에 표기하였다. Table 2에서 Total의 숫자는 RT-qPCR과 RT-ddPCR 각각에서 검출된 시료를 숫자이다. 현장 시료에서 두 분석을 수행한 결과, 고추 현장 시료 45개 중 RT-qPCR 분석에서는 16개의 현장 시료에서 TSWV가 검출되었고, RT-ddPCR에서는 18개 시료에서 TSWV가 검출되었다. 두 방법에서 공통적으로 분석된 시료 수(RT-qPCR, positive; RT-ddPCR, positive)는 13개가 검출되었고, RT-qPCR에서 검출 안된 29개의 시료 중 5개의 시료(RT-qPCR, negative; RT-ddPCR, positive)가 RT-ddPCR를 통해 검출되었다(Table 2). 이러한 결과는 RT-ddPCR 분석이 RT-qPCR 분석보다 높은 정확도와 민감도를 갖는 것이 확인되었으며, 낮은 농도의 TSWV 감염 시료에서의 정량적 검출을 더 신뢰할 수 있고 TSWV 감염 원예작물의 초기 진단에 유용하게 사용될 수 있다.
Acknowledgments
This work was carried out with the support of Cooperative Research Program for Agriculture Science and Technology Development (Project No. PJ014947032021) Rural Development Administration, Republic of Korea.
Fig. 2.
Fluorescence amplitude plot of the annealing temperature gradient for the reverse transcription droplet digital polymerase chain reaction (RT-ddPCR) assay. The Blue dots above the pink horizontal line (threshold) represents positive PCR amplification droplets for tomato spotted wilt virus and gray dots represent negative droplets. The X and Y axes represent the number of droplets and fluorescence signal amplitude, respectively. Each RT-ddPCR reactions are divided by a yellow vertical dotted lines and has an annealing temperature gradient ranging from 48°C to 62°C.
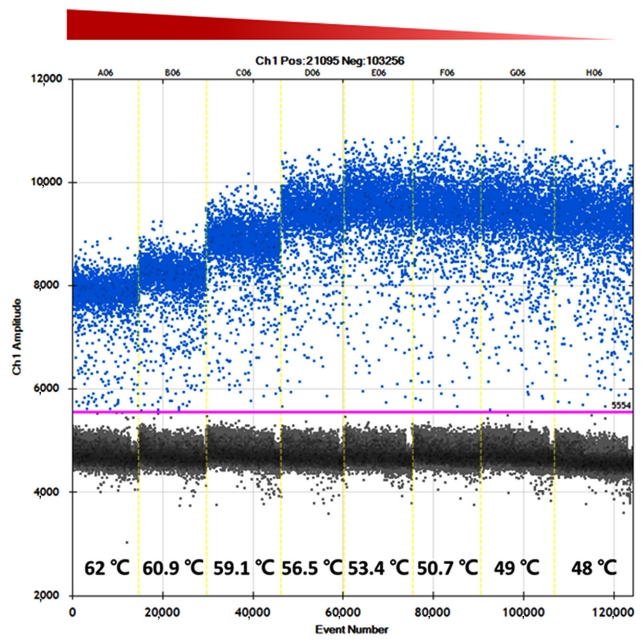
Fig. 3.
Specificity and cross-reactivity testing of reverse transcription droplet digital polymerase chain reaction assay for detection of tomato spotted wilt virus (TSWV). Specificity was confirmed in cucumber mosaic virus (CMV), pepper mottle virus (PepMoV), pepper mild mottle virus (PMMoV), broad bean wilt virus 2 (BBWV 2), healthy leaf (N, negative control) and the negative template control (NTC).
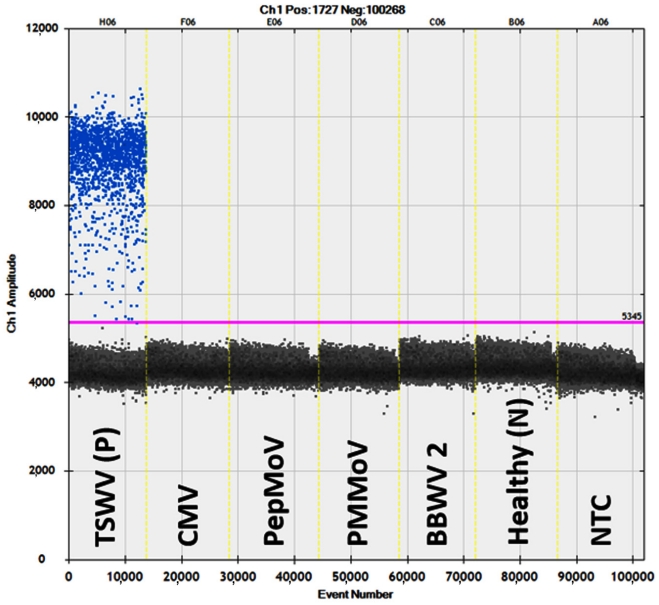
Fig. 4.
Linear regression of assay results determined using serial dilutions of tomato spotted wilt virus (TSWV)-cDNA. (A) Reverse-transcription quantitative real-time polymerase chain reaction (RT-qPCR) and (B) reverse transcription droplet digital polymerase chain reaction (RT-ddPCR) analyses of serial dilutions of the TSWV-cDNA. Quantitative correlation was performed by plotting the quantification cycle of RT-qPCR and the logarithm of the measured concentration of RT-ddPCR against the logarithm of the initial concentration. The correlations of quantitative linearity for RT-qPCR and RT-ddPCR are R2=0.9998 and R2=0.9935, respectively.
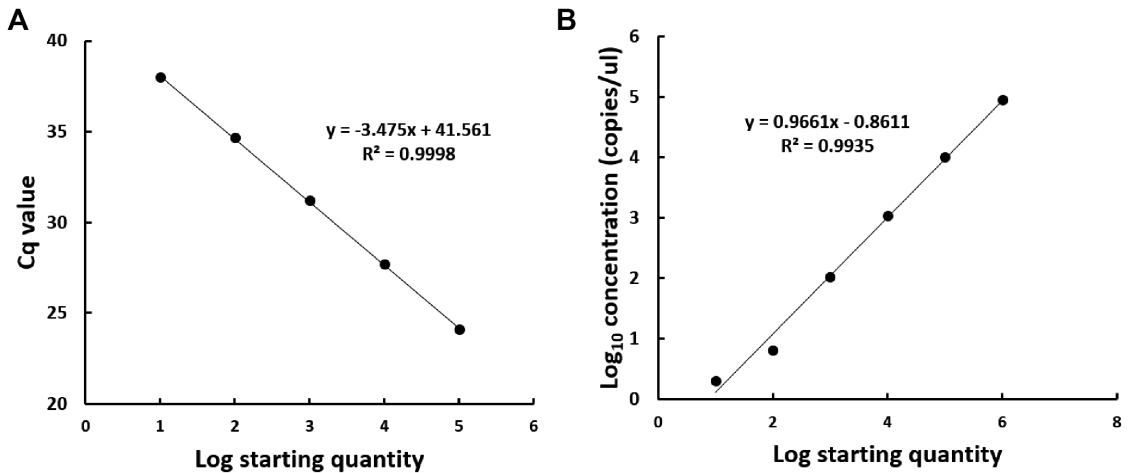
Table 1.
Sensitivities of the RT-qPCR and RT-ddPCR assays for TSWV
References
Ali, M. E., Hashim, U., Mustafa, S., Man, Y. B. C., Dhahi, T. S., Kashif, M. et al. 2012. Analysis of pork adulteration in commercial meatballs targeting porcine-specific mitochondrial cytochrome b gene by TaqMan probe real-time polymerase chain reaction. Meat Sci. 91: 454-459.


Bahder, B. W., Zalom, F. G., Jayanth, M. and Sudarshana, M. R. 2016. Phylogeny of geminivirus coat protein sequences and digital PCR aid in identifying Spissistilus festinus as a vector of grapevine red blotch-associated virus. Phytopathology 106: 1223-1230.


Cho, J.-D., Kim, J.-S., Kim, J.-Y., Kim, J.-H., Lee, S.-H., Choi, G.-S. et al. 2005. Occurrence and symptoms of tomato spotted wilt virus on vegetables in Korea (I). Res. Plant Dis. 11: 213-216.

Cho, J. J., Mau, R. F. L., Hamasaki, R. T. and Gonsalves, D. 1988. Detection of tomato spotted wilt virus in individual thrips by enzyme-linked immunosorbent assay. Phytopathology 78: 1348-1352.

Debreczeni, D. E., Ruiz-Ruiz, S., Aramburu, J., López, C., Belliure, B., Galipienso, L. et al. 2011. Detection, discrimination and absolute quantitation of tomato spotted wilt virus isolates using real time RT-PCR with TaqMan MGB probes. J. Virol. Methods 176: 32-37.


Dube, S., Qin, J. and Ramakrishnan, R. 2008. Mathematical analysis of copy number variation in a DNA sample using digital PCR on a nanofluidic device. PLoS ONE 3: e2876.



Fan, H. C. and Quake, S. R. 2007. Detection of aneuploidy with digital polymerase chain reaction. Anal. Chem. 79: 7576-7579.


Floren, C., Wiedemann, I., Brenig, B., Schütz, E. and Beck, J. 2015. Species identification and quantification in meat and meat products using droplet digital PCR (ddPCR). Food Chem. 173: 1054-1058.


Fukuta, S., Ohishi, K., Yoshida, K., Mizukami, Y., Ishida, A. and Kanbe, M. 2004. Development of immunocapture reverse transcription loop-mediated isothermal amplification for the detection of tomato spotted wilt virus from chrysanthemum. J. Virol. Methods 121: 49-55.


Gutiérrez-Aguirre, I., Rački, N., Dreo, T. and Ravnikar, M. 2015. Droplet digital PCR for absolute quantification of pathogens. Plant Pathology: Methods in Molecular Biology. Vol. 1302, ed Lacomme, C.pp. 331-347. Humana Press, New York, NY, USA.
Han, J.-H., Choi, H.-S., Lee, J., Kim, J.-D., Lee, W. P., Choi, H.-S. et al. 2012. Screening of tomato spotted wilt virus resistance in tomato accessions. Korean J. Hortic. Sci. Technol. 30: 171-177.

Han, J.-H., Lee, W. P., Lee, J., Kim, M.-K., Choi, H.-S. and Yoon, J. B. 2011. Symptom and resistance of cultivated and wild Capsicum accessions to tomato spotted wilt virus. Res. Plant Dis. 17: 59-65.

Higuchi, R., Fockler, C., Dollinger, G. and Watson, R. 1993. Kinetic PCR analysis: real-time monitoring of DNA amplification reactions. Bio/Technology 11: 1026-1030.

Hindson, B. J., Ness, K. D., Masquelier, D. A., Belgrader, P., Heredia, N. J., Makarewicz, A. J. et al. 2011. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal. Chem. 83: 8604-8610.



Hull, R. 2013. Plant Virology. Academic Press, San Diego, CA, USA. 1118 pp.
Karavina, C. and Gubba, A. 2017. Detection and characterization oftomato spotted wilt virus infecting field and greenhouse-grown crops in Zimbabwe. Eur. J. Plant Pathol. 149: 933-944.

Kim, J.-H., Choi, G.-S., Kim, J.-S. and Choi, J.-K. 2004. Characterization of tomato spotted wilt virus from paprika in Korea. Plant Pathol. J. 20: 297-301.

Kurstak, E. 1981. Handbook of Plant Virus Infections: Comparative Diagnosis.. Elsevier, Amsterdam, The Netherlands. 944 pp.
Lee, H.-J., Cho, I.-S., Ju, H.-J. and Jeong, R.-D. 2021a. Development of a reverse transcription droplet digital PCR assay for sensitive detection of peach latent mosaic viroid. Mol. Cell. Probes. 58: 101746.


Lee, H.-J., Cho, I.-S., Ju, H.-J. and Jeong, R.-D. 2021b. Rapid and visual detection of tomato spotted wilt virus using recombinase polymerase amplification combined with lateral flow strips. Mol. Cell. Probes. 57: 101727.


Liu, Y., Wang, Y., Wang, Q., Zhang, Y., Shen, W., Li, R. et al. 2019. Development of a sensitive and reliable reverse transcription droplet digital PCR assay for the detection of citrus yellow vein clearing virus. Arch. Virol. 164: 691-697.


Mehle, N., Dobnik, D., Ravnikar, M. and Novak, M. P. 2018. Validated reverse transcription droplet digital PCR serves as a higher order method for absolute quantification of potato virus Y strains. Anal. Bioanal. Chem. 410: 3815-3825.


Morrison, T., Hurley, J., Garcia, J., Yoder, K., Katz, A., Roberts, D. et al. 2006. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34: e123.



Nakano, M., Komatsu, J., Matsuura, S.-I., Takashima, K., Katsura, S. and Mizuno, A. 2003. Single-molecule PCR using water-in-oil emulsion. J. Biotechnol. 102: 117-124.


Oetting, R. 1991. The effect of host species and different plant components on thrips feeding and development. In: Voris-Thrips-Plant Interaction of Tomato Spotted Wilt Virus, Proceedings of a USDA Workshop., eds. by H. Hsu and R. H. Lawson, pp. 15-19. ARS-US Department of Agriculture, Agricultural Research Service, Beltsville, MD, USA.
Ottesen, E. A., Hong, J. W., Quake, S. R. and Leadbetter, J. R. 2006. Microfluidic digital PCR enables multigene analysis of individual environmental bacteria. Science. 314: 1464-1467.


Pandey, B., Mallik, I. and Gudmestad, N. C. 2020. Development and application of a real-time reverse-transcription PCR and droplet digital PCR assays for the direct detection of potato mop top virus in soil. Phytopathology 110: 58-67.


Parrella, G., Gognalons, P., Gebre-Selassiè, K., Vovlas, C. and Marchoux, G. 2003. An update of the host range of tomato spotted wilt virus. J. Plant Pathol. 85: 227-264.
Peters, D., De Avila, A. C., Kitajima, E. W., Resende, R. O., De Haan, P. and Goldbach, R. 1991. An overview of tomato spotted wilt virus. In: Voris-Thrips-Plant Interaction of Tomato Spotted Wilt Virus, Proceedings of a USDA Workshop., eds. by H. Hsu and R. H. Lawson, pp. 1-14. ARS-US Department of Agriculture, Agricultural Research Service, Beltsville, MD, USA.
Pinheiro, L. B., Coleman, V. A., Hindson, C. M., Herrmann, J., Hindson, B. J., Bhat, S. et al. 2012. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal. Chem. 84: 1003-1011.


Roberts, C. A., Dietzgen, R. G., Heelan, L. A. and Maclean, D. J. 2000. Real-time RT-PCR fluorescent detection of tomato spotted wilt virus. J. Virol. Methods 88: 1-8.


Sastry, K. S. and Zitter, T. A. 2014. Plant Virus and Viroid Diseases in the Tropics. Vol. 2. Epidemiology and Management.. Springer Science & Business Media, New York, NY, USA. 489 pp.

- TOOLS
-
METRICS

-
- 0 Crossref
- 0 Scopus
- 2,743 View
- 111 Download
- ORCID iDs
-
Rae-Dong Jeong

https://orcid.org/0000-0002-6370-8909 - Related articles
-
Application of Electrophoretic Methods for differentiation of Trichoderma species1984 June;23(2)




 PDF Links
PDF Links PubReader
PubReader ePub Link
ePub Link Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print