식물바이러스 면역반응 최신 연구 동향 및 전망
Plant Immunity against Viruses: Moving from the Lab to the Field
Article information
Abstract
Plant viruses cause significant yield losses and continuously threaten crop production, representing a serious threat to global food security. Studies on plant-virus interactions have contributed to increase our knowledge on plant immunity mechanism, providing new strategies for crop improvement. The prophylactic managements consist mainly following international legislations, eradication of infected plants, and application of pesticide to decrease the population of vectors. Hence, putting together the pieces of knowledge related to molecular plant immunity to viruses is critical for the control of virus disease in fields. Over the last several decades, the outstanding outcomes of extensive research have been achieved on comprehension of plant immunity to viruses. Although most dominant R genes have been used as natural resistance genes, recessive resistance genes have been deployed in several crops as another efficient strategy to control viruses. In addition, RNA interference also regulates plant immunity and contribute a very efficient antiviral system at the nucleic acid level. This review aims at describing virus disease on crops and summarizes current resistance mechanisms. Furthermore, we will discuss the current biotechnological approaches to control viral diseases and the future questions that are to be addressed to secure crop production against viruses.
서론
절대기생체인 식물바이러스는 기주의 다양한 생리적 기작을 이용하여 복제하고 침입을 한다. 바이러스는 유전적인 측면에서 가장 단순한 구조로 핵산인 DNA 혹은 RNA로 구성되어 있으며 꼭 필요한 소수의 유전자를 만들어 내고 외피단백질(capsid)에 캡슐화(encapsidation) 되어 있다. 기주식물 자체의 물리적 장벽(큐티클, 세포벽) 때문에 바이러스는 식물의 상처 혹은 매개체(해충, 선충, 곰팡이)를 통해 식물세포 안으로 이동 되어진다. 바이러스가 기주세포내로 이동되어지고 탈외피(decapsidation)된 후 바이러스는 번역과 복제를 통해 자손을 만들고 새롭게 만들어진 바이러스는 세포간(cell-to-cell) 이동과 원거리(long-distance) 이동을 하여 전신감염을 일으킨다. 경우에 따라서는 종자를 통해 전반이 되기도 한다. 2012년 ICTV (International Committee on Taxonomy of Viruses)에서는 92종의 식물바이러스를 보고했으며, 이들 중 82종의 식물바이러스는 21개의 서로 다른 과(Family)로 분류되었다(King 등, 2012).
바이러스의 식물체 침입은 다양한 작물에서 생리적 불균형을 야기시키고 이로인해 크나큰 경제적 손실을 야기하고 있다. 농가에서는 바이러스병 저항성 작물과 다양한 바이러스 방제 방법을 이용해 작물을 재배하고 있다. 지난 수십년간 바이러스와 식물간의 상호작용 연구결과를 바탕으로 도출된 중요한 연구 성과물을 바탕으로 새로운 바이러스 방제 방법들이 소개되고 있다. 그럼에도 불구하고 최근들어 식물바이러스 피해는 점점 증가되고 있는 실정이다. 이는 국제화로 인한 무역 다변화와 기후변화 등이 주요한 요인으로 지목되고 있다.
이 리뷰 논문은 1) 주요 식물바이러스 병과 이에 따른 작물 생산량에 미치는 영향에 대한 개요, 2) 현장에서 사용 되고 있는 바이러스 병 방제 전략 요약, 3) 식물바이러스 병 면역반응에 대한 최신 연구동향, 4) 이를 활용한 기술적 바이러스 방제 방법에 대한 전망을 기술하고자 한다.
바이러스에 의한 주요 작물 피해
식물바이러스 병 방제는 농민뿐만 아니라 국가, 식물병리학자들, 소비자들에게도 매우 중요한 문제이다. 바이러스병은 전세계적으로 다양한 작물에서 양적 및 질적으로 큰 피해를 야기하고 있다. 비록 전세계적으로 바이러스 병에 의한 경제적 손실을 정확히 계산하기는 어렵지만, 매년 발생하는 경제적 손실액은 32조 550억원으로 추정된다(Sastry와 Zitter, 2014). 가장 피해를 많이 야기시키는 바이러스 중 Cassava mosaic virus는 매년 아프리카, 인도, 스리랑카에서 2500만톤 이상 경제적 손실을 야기시키며(Calvert와 Tresh, 2002; Legg와 Thresh, 2000;Thresh와 Cooter, 2005). Potato leafroll virus는 매년 미국과 영국에서 감자 손실액이 각각 1000억원과 640억원에 달하고 있다(Sastry와 Zitter, 2014). 오렌지 재배에서는 전세계적으로 Citrus Tristeza virus가 1000억원의 피해를 끼치고 있다(Harper, 2013). 식량작물(보리, 밀, 옥수수 등)에서는 영국에서만 Barley yellow dwarf virus가 128억원의 손실을 야기시키고 있다(Sastry와 Zitter, 2014). 벼 생산량에 피해를 주는 바이러스 병은 동남아시아권에서만 1조 6000억원의 피해를 주고 있다(Sasaya 등, 2013). 또한 1970년부터 현재까지 Plum pox virus는 유럽에서만 12조원에 달하는 피해를 주고 있다(Cambra 등, 2006). 다년생 작물과 과수의 바이러스 피해는 작물의 생산량을 감소시킬 뿐만 아니라 재배 시간 및 비용 손해도 고려되어야 할 것이다(Hull, 2013). 종종 단독 바이러스 감염에서는 병징이 보이지 않아 크게 바이러스 병 피해가 없을 것이라 과소평가되고 있는 바이러스도 있지만 다른 병원균들과 복합감염이 되면서 큰 경제적 손실을 야기시키는 경우도 있다(Hull, 2013). 더욱 중요한 것은 최근 기후변화 등 새롭게 출현하는 병원균 중 50% 이상이 식물바이러스이라는 점이다. 따라서 식물바이러스는 전세계적으로 작물 피해, 국제 식량문제 등 인간 삶에 있어 전반적으로 위협요인으로 예방 및 방제 전략이 필요하다.
식물바이러스 병 예방 및 방제 전략
식물바이러스는 세포내에 서식하는 절대기생체이기 때문에, 기주 감염 후 치료제가 없어 농가에서 직접 방제하기가 매우 어려운 실정이다. 따라서 예방적 차원의 방제법으로 활용이 농가에서 바이러스 병 발생을 억제하는데 중요하다. 경종적 방법, 생물학적 안전조치, 바이러스 매개체 관리 등이 대표적으로 활용되고 있다(Fig. 1). 최근들어 분자생물학 기법 기반 신속·정확한 바이러스 현장진단용 기술이 발전하게 되어 농가에서 꾸준하게 바이러스 존재여부의 모니터링이 가능해졌다(Boonham 등, 2014). 바이러스 진단의 신뢰도는 바이러스 방제에 중요한 지표가 되며, 감염된 농가에서 신속하게 감염된 식물을 제거할 수 있는 기초적인 자료가 되기도 한다. 대부분의 바이러스는 매개체(해충, 선충, 곰팡이 등)들을 통해 전반이 이루어지기 때문에 적절한 살충제를 사용하여 매개체의 수를 줄이면 바이러스의 발병률도 줄어들게 된다. 또한 주요 작물 재배지 근처에 바이러스 비기주 식물을 재배하여 바이러스의 전반을 줄이는 것도 좋은 방법으로 활용되고 있다(Bragard 등, 2013). 바이러스병 발생은 기존의 작물에서부터 새로운 작물 재배로 발생되는 신종 바이러스 혹은 변이체로 문제가 발생되기도 한다. 비록 이와 같은 현상은 바이러스 유전자와 기주인자(host factor)간의 복잡한 진화적 측면에서 야기되는 문제이기도 하지만, 바이러스 병은 병저항성을 유지시킬 수 있는 재배지의 공간구조 및 구획을 적절히 조절하여 방제가 가능하기도 하다(Elena 등, 2011). 또한 국제무역의 다변화 등으로 인해 다양한 바이러스 유입 식물체 조직(종자, 과실, 접목, 대목, 유묘, 꽃 등)으로부터 발생 가능한 피해를 줄이기 위해 바이러스 무병묘 관련 식물위생 국제 조약을 준수하는 것도 중요한 방법 중에 하나이다. 초기에는 단순히 바이러스 병저항성 유전자를 이용한 저항성 품종의 개발 및 사용으로 재배지에서 바이러스 피해를 줄이는데 가장 효과적이고 지속가능하여 육종가들에 의해 많이 활용하였으나, 20세기 중반부터는 바이러스와 식물간의 상호작용 관련 중요한 연구 결과물을 기반으로 저항성 작물개발을 진행하고 있다.
바이러스에 대한 식물면역 반응
바이러스가 식물에 침입을 하면, 식물은 스스로 방어 시간(감염 초기 혹은 말기), 감염 부위(감염 잎 혹은 비감염 조직), 직접적인 바이러스 유래 분자(바이러스 게놈 혹은 바이러스 유전자)들을 표적함으로써 병발생을 억제시킨다(Fig. 2).
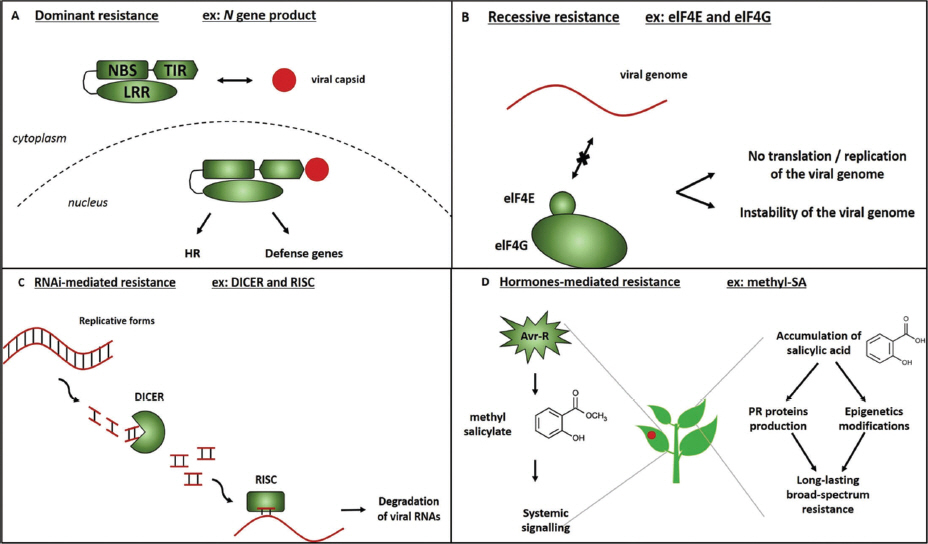
Antiviral immune mechanisms in plants. (A) NBS-LRR R-mediated dominant resistance relies on the interaction between an avirulence factor and a specific R gene, and contributes resistance after the virus infection into the plant for several days. HR-mediated defense response suppresses the virus replication in the infected and neighboring cells. (B) Recessive resistance that is conferred by a recessive gene mutation that encodes a host factor critical for viral infection is a passive and non-inducible resistance. (C) RNA interference (RNAi) targets viral genome. During the replication of viruses, they reach the stage of ssRNA or dsRNA, which trigger host defense responses. (D) Hormones-mediated resistance against viruses is introduced here as antiviral function of salicylic acid (SA) and methyl-salicylic acid (Me-SA) in systemic acquired resistance (SAR).
Dominant resistance (우성유전자 기반 저항성)
식물이 가지는 우성저항성 유전자(dominant resistance gene, R gene)는 식물-바이러스 상호작용 연구를 통해서 특성이 확인 되었으며, 대부분 NBS-LRR (nucleotide binding site leucinerich repeat) 구조를 가지고 있으며, 바이러스의 비기주유전자(avirulence (avr) gene)과 “gene-for-gene” 개념을 통한 특이적 반응 인식을 통해 병저항성이 성립이 된다(Table 1). 이전엔 avr 유전자와 R 유전자가 물리적인 직접적인 상호작용을 통해 병저항성을 유도한다고 생각되어졌지만, 기주내에 다른 기주인자들의 중요성을 강조한 “Guard hypothesis” 모델이 제시되었다(Soosaar 등, 2005). 바이러스에 대한 저항성 단백질인 NBS-LRR은 N-terminal 부분에 TIR (Toll-interleukin-1 receptor) 혹은 CC (Coiled-coil) domain에 따라 다양한 종류로 구분되어 있다(De Ronde 등, 2014; Moffett, 2009). 저항성유전자 기반 바이러스 병 면역반응 기작은 TMV (Tobacco mosaic virus), CMV, TCV(Turnip crinkle virus), PVX (Potato virus X)에 대해 많은 연구가 진행 되어 있다. 바이러스 병 저항성 유전자 중 잘 정리되어 있는 저항성 유전자는 TMV에 대한 담배 N gene이다. N gene은 TIR-NBS-LRR 구조를 가지고 있으며, TMV의 replicase domain과 직접적으로 ATP에 의존하는 상호작용을 가지고 있다(Fig. 2A; Ueda 등, 2006). TMV에 대한 저항성은 기주내 N receptor-interacting protein 1 (NRIP1)이 필요하며, 이 단백질은 chloroplast로부터 세포질과 핵으로 이동하면서 TMV의 replicase 와 N 단백질이 서로 상호작용하는 것으로 알려져 있다(Caplan 등, 2008). PVX에 대한 저항성 유전자인 Rx1 유전자는 전형적인 CC-NBS-LRR 구조를 가지고 있으며, PVX의 coat protein (CP, 외피단백질)과의 인식을 통해 저항성을 나타낸다. Rx1의 CC 도메인은 host factor인 ranGTPase-activating protein 2 (ranGAP2)과 heterodimer를 형성하여 직접적으로 Rx1의 기능에 영향을 미친다(Tameling 등, 2010). 비록 ranGAP2와 PVX CP간의 직접적인 상호작용은 발견되지 않았지만, ranGAP2 단백질은 현재까지 알려지지 않은 다른 host factor를 통해 CP와 간접적으로 상호작용을 하며, 이로인해 단백질 복합체의 conformational change로 인해 Rx1 단백질의 활성을 도와준다(Hao 등, 2013). 또한 TCV가 애기장대 Di-17에 침입을 하게 되면 CC-NBS-LRR 구조를 가지고 있는 HRT 저항성 단백질과 rrt라는 recessive locus에 의해 초기에 Hypersensitive response (HR)이 나타나게 되고 이로인해 저항성이 활성을 띄게된다. TCV에 대한 HRT 저항성 단백질 기반 면역반응에는 Salicylic acid (SA)에 관련 유전자 EDS5 (Enhanced disease susceptibility 5), PAD4 (Phytoalexin deficient 4), SID2 (Salicylic acid induction-deficient 2), SAG101(Senescence-associated gene 101)이 직접적으로 관여하고 있으며, HRT와 EDS1은 직접적인 상호작용을 이루고 있다. 또한, 빛이 TCV 저항성에 직접적으로 영향을 미치는 환경적인 요인이며, 청색광 수용체(blue-light photoreceptor)가 HRT degradation에 영향을 미쳐 TCV에 대한 감수성을 야기시킨다. 특히 최근 RNA silencing 구성 단백질 중 double-stranded RNA-binding protein 4 (DRB4)는 HRT 단백질의 Stability에 영향을 미쳐 면역 반응에 영향을 미치는 것으로 보고 된 바 있다(Jeong 등, 2009; Zhu 등, 2013). CMV Y1에 대한 애기장대 저항성단백질인 RCY1는 HRT와 같은 CC-NBS-LRR 구조를 가지고 있다. CMV의 CP가 Avr factor로 작용하고 있으며, CMV 감염시 HR뿐만 아니라 SA 관련 저항성 유전자인 PR-1과 PR-5의 발현량이 증가한다. TCV-HRT 면역 반응과는 다르게, CMV-RCY1에서는 SA 관련 유전자뿐만 아니라 JA/ethylene 관련 유전자도 함께 발현량이 증가한다. 이는 SA와 JA pathway가 cross-talk 하는 바이러스 면역반응의 대표적인 예시라 할 수 있다(Takahashi 등, 2002).
저항성 유전자의 발현에 의한 면역반응은 칼슘이온유입(calcium ion influx), 맵카이네이즈 신호전달(MAPK signaling), 활성산소(reactive oxygen species) 함량, SA 함량 증가, 광범위한 전자조절인자들의 재프로그램밍이 관여한다. 더구나 저항성 유전자의 발현은 HR 반응과 직접적인 연관이 있으며, 이때 2개의 lipase인 EDS1과 PAD4가 SAG101와 상호작용이 필요하다(Liu 등, 2002; Wagner 등, 2013). 애기장대에서 EDS1/PAD4/SAG101 complex는 HRT 단백질 기반 TCV 저항성을 조절한다(Zhu 등, 2011). 최근 연구에 의하면 R protein 기반 신호전달기작에서 저항성 과정과 HR은 식물면역반응에서 중요한 현상이지만, 서로 다른 생리학적 기작을 따르고 있다 (Bai 등, 2012). TMV-N 과 PVX-Rx1 반응 저항성 단백질의 R protein 기반 면역반응은 국부적으로 발생하지만, 이 반응은 전신으로 면역반응을 유도하는 전신획득저항성(Systemic acquired resistance, SAR)을 일으키기도 한다(Liu 등, 2010). 기존에 알려져 있던 저항성 유전자와는 다르게, Tobacco etch virus (TEV)에 대한 저항성 유전자인 RTM 유전자는 non-NBS-LRR 구조를 가지고 있다. 애기장대에 이들 RTM 유전자들은 TEV, Lettuce mosaic virus virus (LMV), Plum pox virus (PPV) 저항성에 관여되고 있다(Cosson 등, 2012). 이들 RTM 단백질은 체관에 존재하는 복합체 단백질로 장거리 이동하는 바이러스의 이동을 억제함으로써 식물에 저항성을 나타나게 한다(Cosson 등, 2010). 기존의 NBS-LRR 구조를 가지고 있는 저항성 단백질과는 다르게 RTM 기반 저항성은 HR 반응과 SA 함량 증가가 관찰되지 않는다. TMV에 대한 토마토 Non-NBS-LRR 저항성 유전자인 Tm-1은 TMV의 복제단백질과 직접적으로 상호작용하여 바이러스의 복제를 강력하게 방해한다(Ishibashi와 Ishikawa, 2013). 최근들어 고추 유전자인 Pvr9은 담배에서 Potyvirus인 Pepper mottle virus (PepMoV)에 대해 HR에 관여하는 것이 확인되었다. 이 Pvr9 유전자는 고추 염색체 6번에 위치하고 있으며 1298개의 아미노산으로 이루어져 있으며, PepMoV의 NIb 유전자는 Pvr9 기반 HR 유도인자로 확인되었다(Tran 등, 2015). 또한 게놈기반 접근방법을 이용해 고추로부터 Potyvirus와 Tomato spotted wilt virus (TWWV) 저항성 유전자인 Pvr4와 Tsw를 선발하여 식물면역수용체의 다양한 진화에 대해서도 설명한다(Kim 등, 2017).
Recessive resistance (열성유전자 기반 저항성)
식물바이러스 저항성 유전자 200개 중 50%는 열성유전을 가지고 있다(Diaz-Pendon 등, 2004). 이들 열성유전자 기반 저항성 기작은 다른 병원균보다 바이러스에서 더욱더 일반적이다. 이러한 열성유전자는 바이러스 병 저항성 작물 품종 개발에 중요한 방법으로 이용되고 있다. 열성저항성은 종종 바이러스의 완전한 생물학적 병환에 필요한 기주인자의 결여로 발생되는 저항성 기작이다. 지난 수십년간 열성저항성 유전자들이 작물에 도입이 되었으며 이들은 주로 eukaryotic initiation factors (eIFs)인 4E와 4G 유전자들이다(Fig. 2B; Wang과 Krishnaswamy, 2012). 처음에 potyvirus 감염에 효과를 보인 eIF4E와 eIF4G 유전자는 bymoviruses, ccumberviruses, ipomovirus, sobemovirus, carmovirus, waikiviruses 등 다양한 바이러스 저항성 작물개발에 도입이 되었다 (Wang과 Krishnaswamy, 2012). 흥미롭게도 eIF4E 형질전환 시킨 토마토와 감자 작물에서는 potyvirus에 대해 저항성을 나타내기도 했다(Cavatorta 등, 2011). 최근 식물의 translation 관련 인자들, translation initiation factor 4B(eIF4B), translation elongation factors 1A, 1B (eEF1A와 EEF1B), poly(A)-binding proteins (PABPs)들은 애기장대와 담배에서 바이러스 복제에 필요한 인자로 알려져 있다(Hwang 등, 2013;Li 등, 2014b). 이러한 번역관련 인자들은 바이러스방제에 새로운 중요한 재료로 활용될 것으로 생각된다. 감자에서는 다른 열성저항성 유전자인 ra 유전자는 Potato virus A (PVA)의 유관속 이동을 방해하는 기작을 이용해 저항성 작물 개발이 이루어지고 있다. 애기장대에서는 Watermelon mosaic virus에 저항성을 갖게 하는 rwm1는 nucleus-encoded chloroplast phosphoglycerate kinase를 암호화하고 바이러스 침입 초기에 바이러스 복제에 저해에 영향을 미친다(Ouibrahim 등, 2014). 애기장대 돌연변이체 tom1과 tom2A는 TMV의 복제 및 이동이 억제가 된다. 이들 유전자들은 세포막 단백질의 기능을 가지고 있어서 그 기능이 결여된 돌연변이체는 바이러스의 복제가 억제 된다(Ishibashi 등, 2012). 이렇듯 바이러스 복제와 밀접한 관련이 있는 기주 세포막 관련 유전자들을 선발하면 바이러스 저항성 작물 개발에 중요한 인자가 될 것으로 생각되어진다(Barajas 등, 2014; Hyodo 등, 2013).
RNA interference 기반 면역 반응
지난 10년간 RNA interference (RNAi; gene silencing) 기작은 대부분의 진핵생물에 진화적으로 보존되어 있다고 알려져 있었고, 이 현상은 double-stranded RNAs (dsRNAs)에 의한 기작이다. 이들 dsRNAs는 ribonuclease III-type DICER-like (DCL) 효소에 의해 21-24 뉴클리오타이드인 small RNAs (sRNAs)로 잘려지고, 이들은 RNA-induced cytoplasmic silencing complex (RISC)와 ARGONAUTE (AGO) 단백질들과 함께 complex를 이루게된다. RISC에 결합된 sRNAs는 타겟하는 mRNA에 상보적으로 결합하고 그 부분을 절단한다(Bologna와 Voinnet, 2014). 식물에서는 silencing pathway가 다양하게 이루어져 있으며 서로 중복되는 사례가 많다.
대부분의 식물바이러스는 RNA 게놈으로 이루어져 있으며, 바이러스 복제과정 중 바이러스의 RNA-dependent RNA polymerase (RdRp)에 의해 바이러스 게놈이 dsRNA 형태를 가지는 중간 단계를 거치게 된다. 이러한 바이러스 dsRNA는 RNA-silencing 기구에 의해 virus-derived small RNAs (vsRNAs)로 만들어져 결국 바이러스 증식을 억제한다(Pumplin과 Voinnet, 2013; Fig. 2C, 3). vsRNA와 RISC와의 결합은 바이러스 핵산을 특이적으로 분해하고, 이러한 현상은 plasmodesmata와 체관등을 통해 전신으로 이동을 하게 된다. 이러한 과정은 바이러스에 감염되지 않은 잎에 RNA silencing을 유도시키고 식물이 바이러스에 대한 복제를 억제하는 기작으로 알려져 있다. 이러한 현상은 바이러스 특이적 현상이기에 plant adaptive immunity라고도 한다(Voinnet, 2001). DCL4는 RNA virus-derived vs-RNAs를 생성하는데 주요한 단백질이며, 몇몇 경우에는 DCL2가 DCL4의 기능을 대체하기도 한다(Jakubiec 등, 2012; Qu 등, 2008). 이와는 반대로 식물이 가지고 있는 4개의 DCL (DCL1-4)는 DNA virus-derived vsRNAs를 생성한다(Blevins 등, 2011;Moissiard와 Voinnet, 2006). AGO 단백질은 RNase H 효소 기능을 갖는 ribonuclease로 이중형태를 가지고 있는 RNA/DNA를 잘라주는 기능을 한다. 현재까지 식물에서는 10개의 AGO 단백질이 있는 것으로 알려져 있고, 지금까지 AGO1, AGO2, AGO7이 antiviral RNAi 기작에 관여하는 것으로 알려져 있다(Qu 등, 2008; Wang 등, 2011; Zhang 등, 2012). 특히 viruses-derivedvsRNAs은 AGO1과 AGO2에 의한 결합에 의해 주로 이루어지며, 특히 ago1 ago2 돌연변이체에서는 과감수성 현상을 확인할 수가 있다(Wang 등, 2011). 또한 antiviral RNAi는 신호전달 시작과 증폭을 위해 RDR1, RDR2, RDR6가 관여하는 것으로 알려져 있다(Jiang 등, 2012; Qu 등, 2008). 최근에는 TCV에 대한 저항성 단백질인 HRT와 RNA silencing component들 간의 연구결과에서는 RDR6와 DCL4가 HRT기반 저항성에 직접적으로 필요한 단백질로 밝혀졌으며, DCL4와 직접적으로 상호작용하는 Double-stranded RNA binding 4 (DRB4)는 HRT 단백질의 Stability에 관여하고 그로인해 저항성에도 영향을 미치는 것으로 나타났다(Zhu 등, 2013). 흥미롭게도 Tomato yellow leaf curl virus (TYLCV)에 대해 저항성을 갖는 토마토 저항성 유전자인 Ty-1과 Ty-3는 RDR 단백질을 코딩하고 있다(Verlaan 등, 2013).
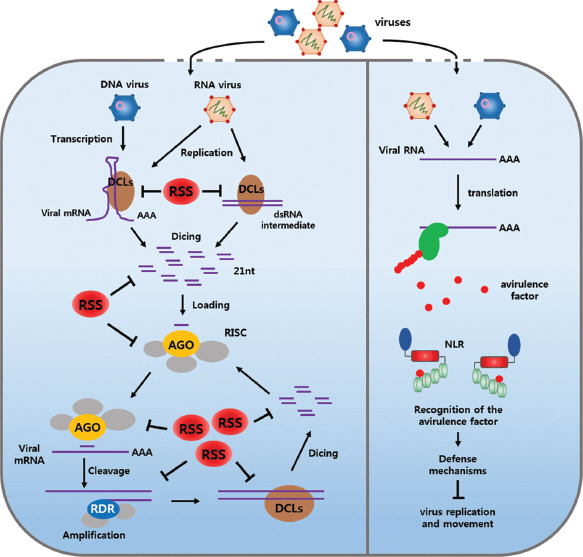
Mechanism of RNAi and R gene-mediated resistance to viruses. Antiviral RNA silencing in plants and its suppression by virus-encoded RNA silencing suppressor (RSSs). RNA silencing is initiated by viral dsRNA or partially double strand hairpins RNAs, which are processed vsiRNAs by Dicers. Following entry into a host cell, viral effectors are expressed from the virus genome. Specific plant R genes interact (directly or indirectly) with these effectors to trigger virus resistance.
바이러스는 식물이 가지고 있는 자연적인 RNA silencing 기작을 피하기 위해 자연스럽게 silencing suppressor 기능을 가지는 유전자를 가지게 되었다. 대부분의 식물바이러스 silencing suppressor는 서로 다른 RNAi 기작에 관여하여 RNAi 기작을 억제하여 바이러스 증식을 가능하게 한다(Bologna와 Voinnet, 2014; Li 등, 2014a). 식물이 가지고 있는 RNAi 기작과 바이러스가 가지고 있는 Silencing suppressor의 관계는 바이러스와 식물간의 끝없는 분자생물학적인 경쟁관계이다(Pumplin과 Voinnet, 2013; Sansregret 등, 2013).
Plant hormone기반 면역 반응
식물호르몬은 식물면역반응에서 중요한 역할을 한다. 식물바이러스가 식물에 침입하면 식물호르몬의 함량과 구성성분이 달라지고, 이와 같은 식물호르몬의 변화가 식물바이러스 공격으로부터 방어를 도와준다(Fig. 4). 지난 수년간 바이러스 침입시 식물면역반응에 관여하는 식물호르몬 선발 및 특성 규명에 대한 연구가 많이 이루어졌다(Table 2; Robert-Seilaniantz 등, 2011). 저항성 유전자 기반 면역반응 동안, 감염된 부위에서부터 세포반응 일어나고 다양한 식물호르몬들의 함량이 증가되어 최대 몇 주 동안 면역반응을 유도하는데 이러한 현상을 SAR이라고 한다(Fig. 2D; Fu와 Dong, 2013).
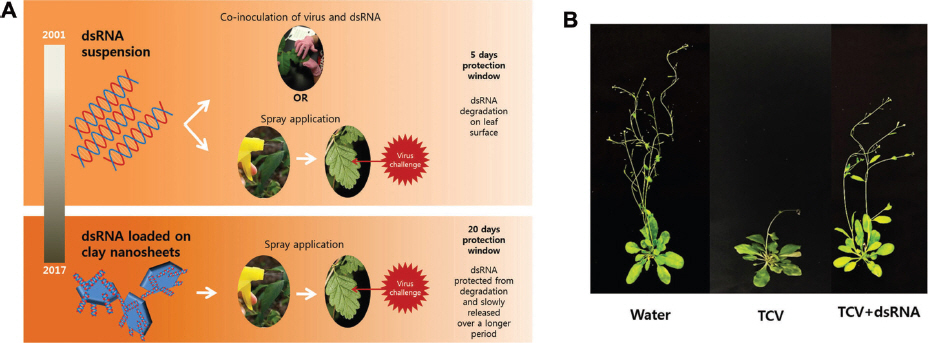
Schematic diagram of exogenous application dsRNA. (A) Top: Naked dsRNA was either inoculated with a homologous virus or dsRNA was sprayed onto the leaf followed by virus inoculation. Double-stranded RNA delivered to the plant cell provides RNAi-mediated protection for up to 5 days postspray. Bottom: Clay nanosheets loaded with dsRNA are sprayed onto the plant and inoculated with the homologous virus. Increased stability triggers efficiency of protection for up to 20 days postspray. (B) Exogenous application of naked dsRNA protects Arabidopsis plants from TCV. Arabidopsis plants were sprayed on day 0 and challenged with TCV on treated leaves at 5 days post-treatment. Leaf crinkled symptoms were observed 14 days postinoculation. Sprayed with water, TCV only, and sprayed with naked dsRNA homologous to CP of TCV.
살리실산(SA)는 바이러스뿐만 아니라 다른 병원균에 대한 병저항성 반응에서 오래동안 연구되어온 호르몬이다. SA는 다양한 Pathogenesis-related (PR) 단백질 발현에 필요한 호르몬이기도 하다. 아직까지 SA는 바이러스에 미치는 영향에 대해서는 많은 연구가 진행되어 있지 않지만, SA는 바이러스 복제, 세포간의 이동 등에 영향을 미치는 것으로 알려져 있다. TMV에 유도된 HR 반응의 경우, 면역반응이 TMV 침입 후 최대 3주동안 지속이 되었다. 어떻게 SAR이 오랫동안 유지되는지 명확히 알지는 못하나 최근들어 DNA methylation과 chromatin 리모델링과 같은 후성유전학적인 변성이 SAR 반응을 유지하는데 중요하다는 연구결과가 보고된 바 있다(Spoel과 Dong, 2012). 바이러스가 침입하는 동안, 오랫동안 지속되고 포괄적인 저항성 반응은 식물내부에서 증가되는 SA 함량이 pathogenesis-related(PR) 단백질의 전사리모델링에 영향을 준다(Yi 등, 2014). 침입부분에 대한 신호전달의 방출은 식물내부의 Methyl-SA가 지방유래물질과 지방수송단백질이 결합하고 체관을 통해 식물 전체로 이동하는 복합체로 순환한다. 그래서 TMV에 감염된 담배식물에서는 MeSA가 SAR 인식 반응에 직접적으로 연관되어 있다(Dempsey와 Klessig 2012). 최근 연구결과에 따르면 SAR에서 모바일 면역 신호는 다양한 인자들 (MeSA, glycerol-3-phosphate, lipid-transfer protein DIR1, pipecolic acid)이 관여한다고 알려져 있다(Dempsey와 Klessig, 2012; Spoel과 Dong, 2012). 따라서 SA가 만들어지지 못하는 식물체에서는 바이러스에 대해 과민성 반응을 보이게 된다. 또한 SA를 분무식으로 살포해주면 식물내부에 있는 SA 생합성 과정을 발현시켜 바이러스에 대한 이동 및 복제를 억제시킨다.
Jasmonic acid (JA) 또한 바이러스 면역반응에 관련되어 있으며, Avr-R 단백질 인식에 의한 HR 반응은 SA와 JA의 모듈에 의해 조절된다. 일반적으로 JA는 바이러스보다 세균, 곰팡이, 곤충, 선충 등에 대해 저항성에 관여하는 호르몬으로 잘 알려져 있다. SA는 바이러스 면역반응에서 positive regulator로써 역할을 하지만, JA에 의한 역할은 아직 완전하게 이해되지 못했다. 가령, JA는 담배식물에서 TMV 감염 잎에서는 Negative regulator로 작용하지만, 감염되지 않은 N. benthamiana 잎에서는 필수 호르몬으로 알려져 있다. 바이러스는 식물호르몬 기작을 타겟함으로써 진화해 왔으며 종종 SA와 JA의 길항 관계로 작용한다(Kazan과 Lyons, 2014). 비록 바이러스와 식물간의 상호작용기작이 완전히 이해되지 않았지만, 최근 연구결과들에 의하면, Abscisic acid (ABA), Ethylene (ET), Brassinosteroids (BR) 식물 호르몬들도 바이러스 면역반응에 직접적으로 관여한다고 보고되었다(Ali 등, 2014; Chen 등, 2013; Seo 등, 2014). 식물바이러스 감염 후 식물호르몬들의 변화는 반복적으로 이루어지고 있다. 또한 병징 발생동안 다양한 식물호르몬들 사이에 cross-talk은 식물면역반응에서는 필수적인 현상이다.
바이러스 방제 방법: 최신 기술 동향
식물-바이러스 연구의 주된 목적은 효과적인 바이러스 방제법을 강구하는데 그 의의가 있다. 오랫동안 전통적인 육종방법인 유전학 기반 저항성 작물 개발이라는 전략을 실행해 왔으나, 빠르게 진화하는 바이러스의 변이로 인해 이들 저항성이 쉽게 무너질 수 있다. 따라서 저항성 작물의 영속성이 필요하여 최근 새로운 육종방법이 소개되고 있으며 최신생명공학기술과 다양한 식물-바이러스 상호작용에 대한 유용한 연구결과를 융합하여 새로운 녹색혁명을 이루고자 하는 연구가 진행되고 있다.
형질전환법(Engineering resistance)
특성 규명이 잘 되어져 있는 여러 저항성 유전자들을 하나의 식물에 도입하는 현상을 “유전자 집적(Gene Pyramiding)”이라고 한다. 이 전략은 식물-바이러스 상호작용에 효과적으로 도입이 되었고, 바이러스에서 유래된 물질(단백질 혹은 핵산)을 타겟하거나 다양한 바이러스 생활사(복제/번역, 세포간 이동, 전신 이동)에 영향을 미치어 새로운 바이러스의 변이체에 의한 저항성 약화를 줄였다(Quenouille 등, 2013). 하나의 식물에 여러 종류의 형질을 가지고 있는 저항성 유전자를 도입하는 일은 어렵지만, gene pyramiding은 식물-바이러스 상호작용에 근거한 병방제에 방법으로서 중요하다. 1980년초 Agrobacterium tumefaciens를 이용한 식물형질전환 기술이 기존의 육종방법을 개선시켰다. 기존의 육종방법과는 다르게, 인위적으로 식물의 게놈에 유용한 유전자를 도입하여 그 형질을 조절하는 유전공학 기술은 다양한 작물의 종 개발 장벽을 극복하여 새로운 품종개발이 가능해졌다. 바이러스 저항성 관련 유전자변형식물은 크게 세가지로 분류되어진다. 첫번째는 바이러스 우성저항성 유전자를 다른 식물에 도입시키는 것이다. 대표적인 예가 다양한 저항성 유전자를 가지과 작물에 도입시킨 것이다(Candresse 등, 2010; Whitham 등, 1996). 두번째 방법은 pathogen-derived resistance (PDR)이라고 불려지고 있는 바이러스의 단백질 혹은 특정한 게놈 부분을 식물체에 도입시켜 바이러스 증식을 억제하는 것이다. 처음에 보고된 연구는 담배식물에 TMV CP를 발현시킨 식물인데, 이는 침입해 오는 바이러스 입자가 세포내에서 분해 기작을 방해한다(Abel 등, 1986). 다른 연구들에서는 바이러스 게놈 일부분(Replicase, Movement 단백질 등) 혹은 비번역 부분을 도입시켜 저항성 작물을 개발하기도 하였다. 바이러스 단백질 기반 저항성은 추후에 RNAi 기작에 기반을 둔 것으로 이해된다(Tenllado 등, 2004). 최근엔 이러한 RNAi 기반 저항성 작물개발은 바이러스 유래 double-stranded, hairpin RNAs, artificial miRNAs 등을 이용해 활용되고 있다(Lemgo 등, 2013; Shimizu 등, 2011;Zhao와 Song, 2014). 국내에서는 GMO 작물에 대한 규제가 심하지만, 해외의 경우, 호박, 파파야, 복숭아, 감자, 콩 등 다양한 GMO 작물을 활용해 바이러스 병 방제 방법으로 활용하고 있다(Bravo-Almonacid 등, 2012; Zagrai 등, 2008). 비록 RNAi 기반 저항성 작물개발이 성공적으로 이루어지고 있지만, GMO 작물의 대한 생태계 미치는 영향등 부정적 측면으로 인해 여러국가에서 심각한 규제를 하고 있다.
Chemical treatments
포장에서 바이러스방제를 위한 치료는 불가능하다. 그러나 화학물질을 이용한 Priming 개념 식물면역 증대 효과를 증가시켜 바이러스에 대한 병방제가 가능하다. 식물호르몬을 인위적으로 식물에 처리해주면 바이러스와 다른 병원균에 대해서 효과적이고 폭넓은 저항성을 가지게 된다. SA 유사물질인 Benzothiadiazol (BTH)는 바이러스를 포함한 다양한 식물병원균에 대한 병저항성 유도 물질로 사용되고 있다(Takeshita 등, 2013; Trejo-Saavedra 등, 2013). 이러한 프라이밍(Priming) 기반 식물면역유도방법은 친환경적이고 자연조건에서 충분히 식물병 방제 방법으로 제시되고 있다. 이러한 긍정적인 효과가 있음에도, 많은 농가에서는 처리량, 작물 종류, 작물의 생육시기, 병원균 밀도, 기후조건, 처리 시간 등의 복잡한 고려조건으로 의해 BTH와 같은 물질을 사용하고 있지 않다(Gozzo와 Faoro, 2013). 그럼에도 불구하고, 화학물질기반 전신획득 저항성은 농가에서 작물이 병저항성을 획득하기 위해 좋은 전략이다.
Plant vaccination
식물백신화는 교차방어(Cross-protection)와 비슷한 개념이다. 이 방법은 약독성을 가지고 있는 Primary 바이러스 취급에 따라 그 효율성이 달라진다. Primary 바이러스는 백신의 기능을 하며, 약독성바이러스와 엔지니어링된 바이러스로 분류되어진다: 약독성바이러스는 같거나 비슷한 병원력을 가지고 있는 바이러스로부터 교차방어를 유도한다(Ziebell과 Carr, 2010; Nishiguchi와 Kobayashi, 2011). 식물백신화를 이용한 예로서 일본에서 Zucchini yellow mosaic potyvirus(ZYMV)의 약독화 바이러스로 CUBIO ZY-02라는 약제로 등록이 되었으며, 농가에서 바이러스방제제로 활용이 되었다(Kosaka 등, 2009). 또한 CTV의 약독바이러스도 Tristeza병 방제에도 활용이 되었다(Lee와 Keremane, 2013). 하지만 모든 바이러스에 대한 약독바이러스를 선발하는 과정이 쉽지는 않다. 이러한 문제점을 해결하기 위해 최근에는 원하는 병원성 바이러스의 게놈 부분을 바이러스 벡터에 클로닝하여 이를 발현시키는 엔지니어링 백신이 개발되었다(Taki 등, 2013; Tamura 등, 2013). Apple latent spherical cheravirus (ALSV)는 토마토, 상추, 호박, 시금치, 콩, 담배 등 초본류와 사과, 체리, 복숭아, 배, 오렌지 등 과수에 아무런 병징없이 감염이 되는 바이러스인데, 이 바이러스에 여러 종류의 바이러스 게놈 부분을 클로닝하여 VIGS 기반 백신화를 시키기도 하였다(Tamura 등, 2013; Taki 등, 2013).
Double-stranded RNA application
RNAi 기반 타겟 RNA의 상동성 있는 RNA를 발현시킨 식물은 바이러스 방제 측면에서 효과적이고 영속적인 장점을 가지고 있다. 하지만, 이러한 형질전환방법은 시간이 오래 걸리고, 고비용이며, 작물마다 효과적인 형질전환 방법이 다르고, GMO로 인한 국가적 규제 등 다양한 단점을 가지고 있었다. 이러한 문제점을 극복하기 위해서 RNAi 기작을 응용하여 바이러스 dsRNA를 식물에 직접 처리하는 방법이 대두되고 있다(Fig. 4). Tenllado 등(2004)은 RNAi 기반 바이러스 dsRNA 처리를 통해 경제적으로 중요한 Pepper mild mottle virus (PMMoV), Alfalfa mosaic virus (AMV), Tobacco etch virus (TEV) 방제 연구결과를 처음으로 제시하였다. PMMoV의 replicase 유전자의 상동성 있는 dsRNA를 혼합하여 N. tabacum cv. Xanthi 또는 Capsicum chinense에 접종하였더니 PMMoV의 병징이 급격하게 줄어드는 것을 확인하였다. 이러한 바이러스 증식의 억제는 local과 systemic 잎 모두에서 관찰되었다. 이 방법은 타겟 바이러스와 dsRNA 사이에 염기서열 특이성과 상동성 있는 염기서열의 최소 길이와 밀접한 관계가 있다. 가령, PMMoV에 대한 dsRNA가 100% 상동성이 있고 0.6-1 kb의 사이즈에서는 완전한 바이러스 억제효과를 보였다. 이러한 바이러스 특이적 dsRNA 적용은 systemic RNAi를 유도하여 바이러스 저항성을 가지게 된다. 하지만 naked dsRNA 적용은 포장에서 최대 5일까지 그 효과를 보이는 단점이 있다. 이러한 naked dsRNA의 안정성의 문제로 드러난 지속성 단점을 극복하기 위해 Layered double hydroxide (LDH) nanosheet를 이용하여 dsRNA를 전달하는 방법이 시도되었다(Fig. 4A; Mitter 등, 2017). LDH nanosheet는 친환경적이고, 비독성이고, 생분해되고, 생석회 기반 복합체이기에 dsRNA를 “BioClay” 형태로 제형화 시켰다. PMMoV와 CMV 상동성 있는 부분의 dsRNA를 BioClay에 도입시켜 담배식물에 처리하게 되면, LDH nanosheet는 식물체 표면에서 천천히 분해가 되면서 PMMoV와 CMV dsRNA가 식물체 표면에 노출된다. 이후 약 30일간에 식물체 표면에 흡착되어 식물체 내부로 스며들어 침입해 오는 바이러스의 증식을 억제한다. 이 방법은 TCV의 CP 부분을 상동성 있는 dsRNA를 처리한 애기장대 식물에서 그 효과가 있음을 확인하였다(Fig. 4B). 아직 정확한 dsRNA의 식물세포내에서의 이동 경로는 명확히 밝혀지지 않았지만 dsRNA가 식물체내로 흡수되고 식물세포내에서 gene silencing을 유도하는 것으로 예상하고 있다. 이러한 나노입자를 활용한 dsRNA delivery system (DDS)는 바이러스뿐만 아니라 다른 병원균 및 해충방제에도 큰 도움이 될 것으로 예상된다. DDS 방법은 하나의 바이러스뿐만 아니라 타겟하는 여러 종의 바이러스를 동시에 방제할 수 있는 이점도 가지고 있어 한 작물에서 다양한 바이러스 증식 억제에도 효과가 있을 것이라 생각된다.
CRISPR/Cas9를 이용한 유전자 교정
Genome engineering(게놈 공학) 기술은 식물 게놈상의 특정 부위 수정을 통해 형질 개선 목적으로 활용되고 있다(Sovova 등, 2016). 게놈공학은 용이함과 재현력의 장점뿐만 아니라, 게놈상의 원하는 부분을 변화시켜 줌으로써 새롭게 형질이 우수해진 품종을 바탕으로 형질전환식물체를 얻을 수가 있다. 이러한 기술의 활용은 기존의 영속적 형질전환식물체 사용의 여러가지 사회적 이슈였던 문제를 회피할 수 있다(Woo 등, 2015; Zhang 등, 2016). 게놈 공학은 게놈상의 원하는 위치에 double stranded breaks (DSBs)를 인위적으로 발생시킨 후 site specific nucleases (SSNs)가 결합하고 특이적 염기서열을 잘라내는 기작을 말한다(Piatek와 Mahfouz, 2017). 주요한 4개의 SSNs가 분류되어 있다:meganucleases, zinc finger nucleases (ZFNs), transcription activator-like effector nucleases (TALENs), clustered regularly interspaced palindromic repeats/CRISPR-associated 9 (CRISPR/Cas9) (Stella와 Montoya, 2016). 이들 기술들은 바이러스 게놈을 직접적 혹은 기주 유전자를 타켓함으로써 바이러스 저항성 작물개발에 사용되고 있다(Table 3). 연구초기에는 ZFN을 이용하여 begomovirus의 복제 결합 부위를 타겟으로 하여 게놈 공학을 시작하였다. 이후 ZFN 방법을 이용하여 TYLCV와 Beet severe curly top virus (BSCTV) 방제를 위한 작물 개발 연구가 진행되었다(Mori 등, 2013; Sera, 2005). 비록 ZFN 방법은 BSCTV와 TYLCV를 효과적으로 억제시키는 좋은 방법이었지만, 이들 바이러스 염기서열의 특이적 전략은 복합감염이 발생하는 포장에서 사용하기엔 어려움이 있었다. TALENs 방법은 begomovirus에 대한 폭넓은 저항성 작물 개발에 시도되었다(Cheng 등, 2015). ZFNs과 TALENs 방법은 효과적인 게놈 공학 기술이지만, 주요한 단점은 타겟하는 게놈 부위에 결합하는 DNA binding protein 기술이 고비용이고 시간이 오래 걸린다(Ceasar 등, 2016). 이러한 제약들이 CRISPR/Cas9 기술이 도입되면서 관련 문제점 줄어들었다(Stella와 Montoya, 2016).
CRISPR/Cas9 기술은 미생물부터 동식물까지 다양한 종에서 게놈 공학을 위한 기법으로 활용되고 있다(Hsu 등, 2014). DNA 바이러스에 대한 연구에서는 CRISPR/Cas9을 이용하여 geminivirus에 대한 저항성 연구가 진행되었다(Fig. 5). CRISPR/Cas9이 발현되고 있는 N. benthamiana 식물에서는 TYLCV, Beet curly top virus (BCTV), Merremia mosaic virus (MeMV), Bean yellow dwarf virus (BeYDV), BSCTV에 대해 저항성을 보였다(Ali 등, 2016; Baltes 등, 2015; Ji 등, 2015). 흥미롭게도 이들 바이러스의 비번역 타겟 부위가 바이러스 억제효과가 급격히 증가되었다. 현재 RNA 바이러스를 타겟으로 절단시키는 CRISPR/Cas9 방법에 대한 연구는 거의 없다. CRISPR/Cas9를 이용하여 RNA 바이러스를 타겟하기 어려운 이유는 Guide RNA-Cas9은 단지 DNA 바이러스를 인식하고 절단하기 때문이다. 차후에 Cas9이 RNA를 인식하고 절단하도록 시스템을 바꿔야 할 것이고 특히 RNA 염기서열을 잘라낼 수 있는 TYPE III-B CRIPSR-Cas 시스템이 도입되어야 할 것이다(Hale 등, 2009). 현재로써는 CRISPR/Cas9을 이용하여 RNA 바이러스 게놈을 타겟하기보다는 바이러스 증식에 관여하는 식물체내 유전자를 조작하여 바이러스 저항성을 만들 수 있다. 가령, eIF4E와 eIF(iso)4E는 RNA 바이러스의 감염에 직접적으로 관여하는 인자이기에 이들 유전자들을 CRISPR/Cas9를 이용하여 조작하게 되면 바이러스 저항성 작물 개발이 용이하다(Sanfacon, 2015). 따라서 RNA 바이러스 저항성 작물 개발을 위해서는 CRISPR/Cas9를 Recessive 저항성에 활용하여야 할 것이다.
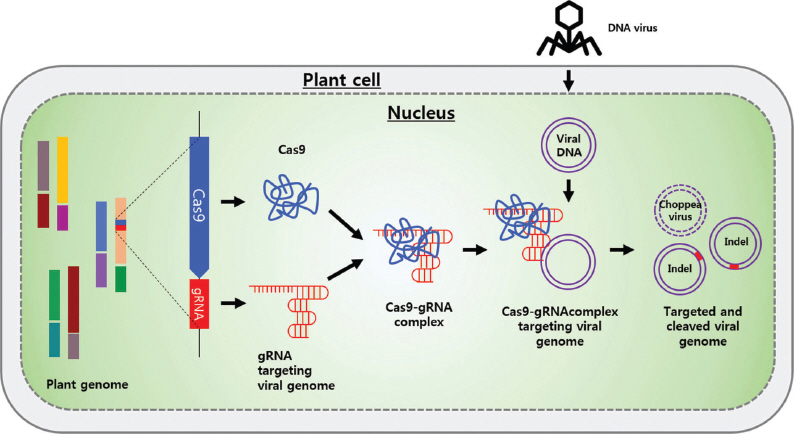
Engineering plants for virus resistance with CRISPR/Cas9 system. Components of the CRISPR/Cas9 machinary, gRNA, and Cas9, are expressed from the plant genome and form gRNA-Cas9 complex. Upon viral infection, the viral DNA replicates through the dsDNA replicative form inside the nucleus of plant cell. The gRNA-Cas9 complex targets the viral DNA dsDNA at complementary target sites and cleaves the viral genome via double stand breaks (DSBs).
결언
최근 국제적 무역 다변화, 재배환경의 변화, 급변하는 기후변화로 인해서 식물바이러스 병 피해가 증가하여 작물을 생산하는데 큰 어려움을 겪고 있다. 바이러스 병 방제 및 관리는 국제적 식량안보와도 큰 연관성이 있다. 현재까지, 바이러스방제 및 작물개발은 바이러스와 식물간의 상호작용 연구결과물을 바탕으로 바이러스학자, 유전학자, 분자생물학자, 육종가들의 노력으로 일구어낸 결실이라고 생각된다. 최근들어 급격하게 발전된 생명공학기술과 식물면역 기작 결과물을 융합하게 되면 돌발바이러스에 대한 피해를 충분히 극복할 수 있을거라 생각된다. 특히 유전자편집기술과 같은 새로운 게놈 공학기술을 기능유전체 연구와 식물면역의 넓은 이해와 접목한다면, 바이러스 병 피해를 감소시킬 수 있는 효과적인 대응 방안으로 활용될 수 있을 것이라 생각된다.
요약
전 세계적으로 주요 작물에서 기후변화, 무역의 다변화 등 여러 요인에 의해 식물바이러스에 의한 작물 생산량 감소 등의 경제적 손실이 심각하다. 이에 경제 작물에서 심각한 바이러스 병피해를 줄이기 위한 여러 바이러스에 대한 광범위한 저항성 작물개발이 시급하다. 식물바이러스 병 예방 및 방제를 위해서 지금까지 연구해왔던 바이러스-식물간의 상호작용 기초 연구결과물 뿐만 아니라 식물면역 관련 과학적 방법 종합화를 통한 응용화된 연구 진행이 필요하다. 본 리뷰에서는 바이러스 저항성 작물 도입을 위해 지금까지 연구되어 왔던 식물면역 기작을 소개하고 이를 활용한 작물 개발 사례를 소개하였다. 또한 유전자교정기술과 같은 게놈 공학 기술을 활용한 바이러스 저항성 작물의 필요성과 연구 방향에 대해 기술하였다. 본 리뷰를 통해 현재까지 알려져 있는 바이러스 면역 기작에 대한 이해를 돕고, 최신 바이러스 병방제 기술들을 소개함으로써 농민들뿐만 아니라 연구자들에게도 도움이 되기를 바라며, 식물면역 연구가 작물 재배 중 발생할 수 있는 바이러스 병 농가 피해를 감소시킬 수 있는 효과적인 대응 방안으로 이어지길 바란다.
Conflicts of Interest
No potential conflict of interest relevant to this article was reported.
Acknowledgement
This work was financially supported by the National Research Foundation of Korea (NRF) grant funded by the Korea government(MSIP) (No. 2017R1C1B1001873) and 2017 Research grant from Kangwon National University.