다양한 작물에서 세균검은점무늬병을 일으키는 Acidovorax valerianellae의 병원성이 다른 2그룹
Two Pathogenic Groups in Acidovorax valerianellae Causing Bacterial Black Spot on the Various Crop Plants
Article information
Abstract
Acidovorax valerianellae had been reported a causal agent of bacterial black spot disease on corn salad in France, 2003 and on watermelon in Korea 2011. In this study, difference in host specificity between 2 groups, corn salad strains and watermelon strains, of Acidovorax valerianellae was recognized and compared. In the pathogenicity test, all 5 watermelon strains showed pathogenicity on the 6 Cucurbitaceae plants but not on corn salad, whereas 4 corn salad strains showed pathogenicity only on the corn salad. Utilization of Biolog substrates was different between watermelon strains and corn salad strains on 4 substrates, Malonic Acid, α- Hydroxybutyric Acid, α-Keto Butyric Acid, and Glycyl-L Glutamic Acid. The phylogenetic tree built with the 16S rDNA sequences showed that all of A. valerianellae stains was grouped into 1 clade separating from the other species of Acidovorax genus. Within A. valerianellae clade, watermelon strains and corn salad strains were separated into 2 sub-groups. REP-PCR analysis also separated the two groups. Host specificity, substrate utilization, and some genetic characteristics suggested that there are two pathogenic groups, watermelon group and corn salad group in A. valerianellae.
서론
프랑스에서 콘샐러드에 발생한 세균검은점무늬병(bacterial black spot)의 병원균으로 A. valerianellae가 2003년에 최초로 보고되었다(Gardan 등, 2003). 아시아에서는 같은 해에 일본에서 A. valerianellae가 차나무(Camellia sinensis)에서 병을 일으킨다고 보고되었고(Tomihama 등, 2006), 그 후 박과식물에서도 세균검은점무늬병의 발생이 보고되었다(Takikawa 등, 2008). 국내에서는 2011년 수박에서 이 병이 발생하였고(Han 등, 2012), 수박의 세균검은점무늬병은 유묘기 초기에는 떡잎에 검은색 점무늬가 나타나고 병이 진전되면서 갈색 반점이 생기고 잎 전체가 검게 변하는 특징을 보이며, 28°C 전후 다습한 조건에서 발생한다(Han 등, 2012). 특히, 이 병은 수박에서 Acidovorax citrulli이 일으키는 과일썩음병(BFB, bacterial fruit blotch)의 병징 및 병원균과 유사하기 때문에 이 2가지 병의 구별이 쉽지 않다. 실제로 과일썩음병의 병징으로 의심되던 병반에서 A. valerianellae이 분리되어 동정이 되는 경우도 있었다 (Song 등, 2015). 병원균인 A. valerianellae는 1970년대에는 비형광성 Pseudomonas sp.로 분류되었고(Rat와 Gardan, 1993), 그 후 일부 비형광성 Pseudomonas는 Acidovorax속으로 분류되었으며(Willems 등, 1990, 1992), 2003년에 A. valerianellae로 재분류 되었다(Gardan 등, 2003). 그람 음성세균인 A. vaelrianellae는 크기가 0.84-0.89×1.54-1.69 μm이고, 한 개의 편모를 가진 막대형 세균이다(Han 등, 2012). King’s B 배지에서는 배양 2일 후 가장자리가 흐릿한 크림색의 콜로니를 형성한다(Grondeau 등, 2007). 병의 전염경로는 토양전염(Grondeau 등, 2003)과 종자전염(Grimault 등, 2006; Grondeau와 Samson, 2009)이 보고되어 있다.
본 연구에서는 국내의 수박에서 분리한 A. valerianellae 균주의 병원성이 기주식물에 따라 콘샐러드로부터 분리된 균주의 병원성과 다름을 인지하고, 수박 분리균주와 콘샐러드 분리균주의 병원성, 표현형 및 유전적 특성을 비교하여 A. valerianellae에 기주에 따라 병원성이 다른 2가지 그룹이 있음을 확인하였다.
재료 및 방법
사용 균주 및 세균 DNA 분리
본 연구에 총 9개의 A. valerianellae 균주들이 사용되었다. 농촌진흥청 농업유전자원센터 미생물은행(Korean Agricultural Culture Collection, KACC, Wanju, Korea)에서 수박분리 1균주, ㈜농우바이오(Nongwoobio. Co., Ltd., Yeoju, Korea)에서 수집한 수박분리 4균주, 그리고 농림수산검역검사본부의 금지품국가수입허가절차를 거쳐 프랑스 균주은행(Collection Francaise de Bacteries Phytopathogenes, CFBP, Beaucouze, France)으로부터 콘샐러드(corn salad)로부터 분리한 4균주를 분양을 받아 사용하였다 (Table 1).
사용된 균주들은 King’s B 액체배지(Peptone 20.0 g, MgSO4·7H2O 1.5 g, K2HPO4 1.5 g/l)에 글리세롤을 15%가 되도록 첨가한 세균 보관용 용액에 현탁하여 -80°C에서 보관하여 사용하였다. 세균 균주들을 28°C에서 48시간 동안 King’s B 배지에 배양한 후 자동핵산분리기(ROCHE, Basel, Switzerland)를 이용하여 DNA를 분리하였으며, 분리된 DNA는 10 ng/µl로 정량한 후 -20°C에 보관하며 사용하였다.
Acidovorax valerianellae 균주별 병원성 조사
분리기주에 따른 A. valerianellae 균주별 박과식물과 콘샐러드에 대한 병원성의 차이를 알아보고자 A. valerianellae 9균주를 박과 8가지 작물인 수박(Citrullus lanatus cv. bravoggul), 멜론(Cucumis melo cv. earthtalent), 오이(Cucumis sativus cv. salim), 참외(Cucumis melo cv. chamsarang), 애호박(Cucurbita moschata cv. nongwooae), 쥬키니호박(Cucurbita pepo cv. sunnyhouse), 대목용 호박(Benincasa cerifera cv. rspower), 대목용 박(Lagenaria leucantha cv. shinhwachangzo)과 콘샐러드(Valerianella locusta)의 유묘에 접종하였다. 각 식물의 종자를 원예용 상토 식물세계® (Nongwoobio. Co., Ltd., Yeoju, Korea)가 담긴 50구 연결 포트에 파종하여 온실에서 재배하였으며, 본엽 1엽이 완전히 전개된 유묘에 병원균을 접종하였다. 병원균들은 King’s B 배지에 도말하여 28°C에서 48시간 배양시킨 후 멸균수에 현탁하고, 현탁액의 흡광도를 분광광도계(Mecasys, Deajeon, Korea)로 OD600nm에서 0.1 (2×108 cfu/ml)로 조절하였다. 이 병원균 현탁액을 유묘에 분무 접종하고, 접종한 식물체를 28°C 생장실 (상대습도 95%)에서 24시간 보관한 후 온실(24-30°C)로 옮겨 관리하였다. 접종 후 3주까지 병징의 변화를 관찰하며 발병주율을 조사하였다. 각 처리 별 50주 씩 3반복으로 실험하였으며, 결과는 SAS (SAS Institute, Inc., Cary, NC, USA) 프로그램으로 DMRT (Duncan’s multiple range test) 분석하였다.
Biolog 기질이용성 조사
분리기주에 따른 A. valerianellae 균주별 기질이용성의 차이를 Biolog GN2 Microplate (Biolog Inc., Haywood, CA, USA)를 이용하여 조사하였다. 수박으로부터 분리된 3균주와 콘샐러드로부터 분리된 2균주를 BUG (Biolog Universal Growth) agar에 스트리크(streak)하여 30°C에서 24 시간 배양하였다. GN/GP Inoculating Fluid에 600 nm에서 52% T 농도가 되도록 현탁하였고, 이 현탁액을 Biolog plate의 각 well에 150 µl씩 분주하여 30°C에서 24시간 동안 배양한 후 변색에 따라 그 기질이용 여부를 조사하였다.
REP-PCR 분석
국내의 수박에서 분리된 A. valerianellae 5균주와 콘샐러드에서 분리된 A. valerianellae 4균주의 유전적 다양성을 비교하기 위하여 REP-PCR을 실시하였다(Louws 등, 1998). 프라이머는 bacterial repetitive elements 중 ERIC sequence를 기반으로 만든 ERIC 2[5’-AAG TAA GTG ACT GGG GTG AGC G-3’]와 ERIC 1R[5’-ATG TAA GCT CCT GGG GAT TCA C-3’]을 사용하였으며, Ex Taq PCR kit (GENETBIO, Daejeon, Korea)를 이용하여 세균의 genomic DNA (gDNA) 10 ng를 포함한 반응액 20 µl로 PCR을 수행하였다. PCR은 95°C에서 7분간 초기 denaturation 과정을 거친 후에 94°C에서 1분, 52°C에서 1분, 65°C에서 8분간의 반응을 30 cycles 거친 후 최종 extension 과정을 65°C에서 16분간 반응시켰다. PCR 증폭산물은 1.5% agarose gel에서 전기영동하여 PCR 결과를 확인하였다.
16S rDNA의 염기서열에 따른 유연관계 분석
수박으로부터 분리한 A. valerianellae 5균주와 콘샐러드로부터 분리한 A. valerianellae 4균주를 포함하여 Acidovorax속 균주들 사이의 16S rDNA 염기서열을 비교하였다. 본 연구에 사용한 A. valerianellae 9균주의 16S rRNA 유전자 염기서열은 universal primer 27F와 1492R을 이용한 PCR로 증폭된 약 1,260 bp의 DNA를 마크로젠(Seoul, Korea)에 의뢰하여 염기서열을 얻었다. NCBI에서 유전자 염기서열 검색을 통해 A. citrulli, A. avenae, A. anthurii, A. caeni 등의 균주들의 16S rDNA 염기서열을 수집하여 본 연구에서 얻은 A. valerianellae 9균주의 16S rDNA 염기서열들과 비교 분석하였다. 본 연구에서 얻은 A. valerianellae 9균주들의 16S rDNA 염기서열과 NCBI에 등록된 다른 대조균주의 16S rDNA 유전자 염기서열의 상동성은 Mega (version 5.05, Pennsylvania, USA)와 BioEdit (version 7.0.1, Tom Hall, Carlsbad, CA, US) 프로그램을 이용하여 분석하였다. 각각의 염기서열은 NCBI의 GenBank에 등록된 서열 중 가장 유사도가 높은 서열과 비교하였으며, CLUSTAL W 프로그램을 이용하여 다중정열 하였다(Thompson 1994). 정열 된 각 염기서열의 evolutionary distance matrix는 K2P (Kimura 2-parameter)로 구하였고, phylogenetic tree algorithm은 MLE (Maximum Likelihood)을 이용하여 계통도를 그렸으며, 지지도를 확인하기 위하여 1,000 번의 부트스트랩(bootstrap)을 수행하여 500 이상의 값을 표기하였다.
결과
Acidovorax valerianellae 균주별 병원성 차이
수박으로부터 분리된 5균주와 콘샐러드로부터 분리된 4균주를 박과 8가지 작물(수박, 오이, 참외, 멜론, 쥬키니호박, 애호박, 대목용 박, 대목용 호박)과 콘샐러드 유묘에 접종하여 기주식물별 발병주율의 차이를 비교한 결과, 수박으로부터 분리된 4균주(NWB SC185, NWB SC186, NWB SC189, NWB SC190)는 박과 8가지 작물 중 6가지 작물인 오이, 수박, 대목용 박, 대목용 호박, 쥬키니 호박, 애호박에서 접종 2일 후부터 수침상이 나타나기 시작하여 5일 후부터는 엽맥을 따라 부정형의 병반을 형성하며 병징이 진전되었다. 병 발생은 접종 3주 후까지 관찰하였으나, 발병주율은 접종 7일 이후에는 변화가 없었다. 박과의 나머지 2작물 중 멜론에서는 4% 미만의 발병주율을 보여주었고, 참외에서는 전혀 병이 발생하지 않았다(Table 2, Fig. 1). 수박으로부터 분리된 나머지 1균주(KACC16997)도 박과식물 중 참외를 제외한 7가지 작물에서 병원성을 보여 주었는데, 수박, 쥬키니호박, 애호박, 대목용 호박에서는 95% 이상의 식물에서 병을 발생시켰으나, 오이(33%), 멜론(7%)에서는 상대적으로 낮은 병원성을 보여주었다. 콘샐러드로부터 분리된 4균주는 콘샐러드에서만 접종 4일 후부터 수침상 병반이 나타나며 병을 일으켰으나, 접종한 박과의 8가지 작물에서는 병을 일으키지 못하였다(Table 2, Fig. 2).

Disease incidence on 8 Cucurbitaceae plants and corn salad inoculated with different strains of Acidovorax valerianellae

Symptoms on cotyledons of Cucurbitaceae and corn salad plants inoculated with Acidovorax valerianellae NWB SC190, a watermelon isolate, at 7 days after inoculation.

Symptoms on cotyledons of Cucurbitaceae and corn salad plants inoculated with Acidovorax valerianellae CFBP4730, a corn salad isolate, at 7 days after inoculation.
이상의 결과를 종합하면, 수박으로부터 분리한 A. valerianellae 균주들은 참외와 멜론을 제외한 6가지 박과식물에서는 높은 발병주율을 보여주었지만, 콘샐러드에서는 전혀 병을 일으키지 못하였으며, 반면에 콘샐러드로부터 분리한 A. valerianellae 4균주는 콘샐러드에서는 높은 발병주율을 보여주었지만 8가지 박과식물에서는 전혀 병을 일으키지 못하였다. 이 결과는 분리된 기주식물에 따른 A. valerianellae 균주별로 기주식물에 대한 병원성에 큰 차이가 있음을 보여주는 결과이다.
기질이용성 차이
분리된 기주식물에 따른 A. valerianellae 균주별로 기질이용성의 차이를 Biolog GN2 Microplate (BIOLOG)를 이용하여 조사하였다. 수박으로부터 분리된 3균주(NWB SC185, NWB SC190, KACC16997)와 콘샐러드로부터 분리된 2 균주(CFBP4723, CFBP6480)의 기질이용성을 비교한 결과, 95개의 기질 중에서 29개(Tween 20, Tween 80, D-Fructose, Pyruvic Acid Methyl Ester, Succinic Acid Mono-Methyl-Ester, Acetic Acid, D-Gluconic Acid, β-Hydroxybutyric Acid, α-Keto Glutaric Acid, α-Keto Valeric Acid, D,L-Lactic Acid, Propionic Acid, Sebacic Acid, Succinic Acid, Bromosuccinic Acid, Succinamic Acid, D-Alanine, L-Alanine, L-Alanylglycine, L-Asparagine, LAspartic Acid, L-Glutamic Acid, L-Leucine, LPhenylalanine, L-Proline, L-Pyroglutamic Acid, L-Serine, Urocanic Acid, Glycerol)에 대해서 수박 및 콘샐러드로부터 분리된 5균주 모두 이용하는 것(양성 반응)으로 나타났으며, 8개 기질(L-Arabinose, γ-Hydroxybutyric Acid, α-Keto Butyric Acid, Malonic Acid, L-Histidine, Glycyl-LGlutamic Acid, L-Threonine, γ-Amino Butyric Acid)에 대해서는 5균주별 이용성의 차이를 보였으며, 수박으로부터 분리한 3균주와 콘샐러드로부터 분리한 2균주 사이에서는 4개 기질(Malonic Acid, α-Hydroxybutyric Acid, α-Keto Butyric Acid, Glycyl-LGlutamic Acid)의 이용성 차이를 보였다(Table 3).
유전적 특성 차이
분리기주가 다른 A. valerianellae 9균주의 유전적 다양성을 비교하기 위해 ERIC 2/ERIC 1R 프라이머를 이용한 REP-PCR을 실시하였다. REP-PCR 결과 수박으로부터 분리된 A. valerianellae 4균주는 서로 동일한 DNA 밴드패턴을, 그리고 콘샐러드로부터 분리된 A. valerianellae 4균주는 서로 동일한 DNA 밴드패턴을 가진 것으로 나타났다. 수박으로부터 분리된 A. valerianellae KACC16997 균주를 제외하면 REP-PCR에 의한 DNA 밴드패턴은 분리된 기주식물별로 두 개의 그룹으로 나누어지는 것을 확인하였다. 수박으로부터 분리된 A. valerianellae KACC16997 균주는 위의 두 그룹과는 다른 패턴을 가진 것으로 나타냈다(Fig. 3).
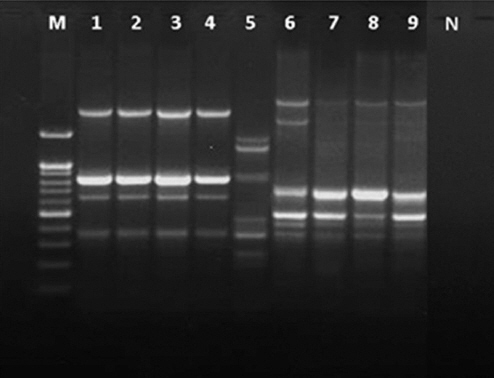
PCR result with primer ERIC 2/ERIC 1R and genomic DNA of Acidovorax valerianellae strains. Lanes 1-9, A. valerianellae NWB SC185, NWB SC186, NWB SC189, NWB SC190, KACC16997, CFBP4723, CFBP4730, CFBP6478, CFBP6480. N was water as negative control.
분리기주가 다른 A. valerianellae 9균주의 16S rDNA 염기서열을 이용하여 계통수(phylogenetic tree)을 작성하여 균주 간 유연관계를 비교하였다. 계통수 작성에는 본 연구에서 사용한 A. valerianellae 9균주의 rDNA 염기서열 외에 NCBI GenBank에 등록된 A. valerianellae 그리고 A. valerianellae 유사한 균주의 16S rDNA 염기서열을 포함시켰다. 계통수 작성에 사용한 모든 A. valerianellae 균주는 Acidovorax의 다른 종과는 다른 clade로 그룹화 되었다(Fig. 4). 그리고 A. valerianellae clade 내에서 수박 분리 A. valerianellae 균주와 콘샐러드 분리 A. valerianellae 균주는 다른 소그룹으로 나뉘어졌다. 16S rDNA 염기서열에서 수박과 콘샐러드로부터 분리된 A. valerianellae 균주 그룹 사이에는 3개의 염기에서 차이가 있었다(Fig. 5). 또한 콘샐러드로부터 분리된 A. valerianellae 4 균주 중에서 CFBP4723, CFBP4730와 CFBP6478, CFBP6480는 서로 다른 clade로 그룹 되었는데 (Fig. 4), 이들 균주들 사이에는 염기서열에서 1개의 차이가 있었다(Fig. 5).

Phylogenetic tree with 16S rDNA sequences of Acidovorax spp. and Acidovorax valerianellae strains. The 16S rDNA sequence of Pseudomonas testosteroni ATCC11996 was used as outgroup.
고찰
본 연구결과 세균검은점무늬병(bacterial black spot)을 일으키는 병원균 A. valerianellae 집단 내에는 기주식물에 따라 병원성이 다른 그룹이 존재하는 것으로 나타났다. 즉 기주식물별 병원성검정 결과 수박 분리 5균주는 참외와 멜론을 제외한 6가지 박과식물에서 높은 병원성(발병주율)을 보여주었지만 콘샐러드에서는 전혀 병을 일으키지 못하였다. 콘샐러드 분리균은 접종한 박과식물에서는 전혀 병을 일으키지 못했지만 콘샐러드에서는 90% 이상의 높은 발병주율을 보여주었다. 특히, 수박분리 균주 중에서도 KACC1997는 다른 수박분리균주와 다르게 오이에서 33%의 발병주율을 보여주었는데, 이는 REP-PCR 결과, 수박 분리균주 내에서도 KACC1997이 다른 패턴을 보여 박과 작물 내 기주별 병원성의 차이와 상관관계가 있을 것으로 보인다.
기질이용성에서도 수박 분리균주들과 콘샐러드 분리균주들 사이에는 4개의 기질에서 차이가 있는 것으로 나타났다. 유전적 분석에서는 16S rDNA 염기서열을 이용한 계통수(phylogenetic tree)는 본 연구에서 사용한 9개의 A. valerianellae 균주들을 한 개의 clade로 그룹화하여 이들 균주들이 동일한 종에 속함을 확인할 수 있었다. 그런데 이 clade 내에서 수박 분리 균주들과 콘샐러드 분리균주들은 다른 그룹으로 묶였고, 실제로 16S rDNA 염기서열에서 이들 2 그룹 사이에는 3개의 염기 서열에서 차이가 있었다. 이상의 결과들은 세균검은점무늬병 (bacterial black spot)을 일으키는 병원균 A. valerianellae 집단내에는 기주에 따른 병원성과 유전적으로 다른 그룹이 존재함을 제시하고 있다.
동일한 종에 속하는 세균의 균주들 사이에 기주식물에 따라 병원성이 다른 경우는 식물병원 세균 집단에서 흔히 존재하는 현상으로 병원형(pathovar)이 그 대표적인 예이다. Pseudomonas syringae는 40개가 넘는 병원형을 가지고 있다(Young, 2010). 앞으로 더 많은 균주를 수집하여 각 균주의 기주특이성과 유전적 다양성의 분석이 필요하겠지만 본 연구결과에서 보여준 수박 분리균주와 콘샐러드 분리균주들 사이의 기주식물에 따른 병원성의 차이는 A. valerianellae 종의 병원균 집단 내에 병원성의 유형의 다른 집단이 존재함을 암시하고 있다.
요약
Acidovorax valerianellae는 콘샐러드에서 세균검은점무늬병 (bacterial black spot)을 일으키는 병원균으로 2003년 프랑스에서 최초로 보고되었고, 2011년에 국내의 수박에서 동일한 병을 일으키는 병원균으로 보고되었다. 본 연구에서는 콘샐러드 분리균주와 수박 분리균주의 2 그룹 사이에 기주특이성을 확인하고 비교하였다. 병원성검정 결과 수박 분리 5균주는 박과 6가지 작물에서 병원성을 보였으나 콘샐러드에서는 병원성이 없었고, 콘샐러드 분리 4균주는 콘샐러드에서만 병원성을 나타내었다. Biolog 기질이용성에서는 총 95개 기질 중 4개(Malonic Acid, α-Hydroxybutyric Acid, α-Keto Butyric Acid, Glycyl-LGlutamic Acid)의 기질에서 차이를 나타냈다. 16S rDNA 염기 서열을 이용하여 계통수를 작성하고 균주 간 유연관계를 분석한 결과, 모든 A. valerianellae 균주는 Acidovorax속의 다른 종과는 별개의 한 개의 clade로 그룹화되었다. 그 clade 내에서 수박 분리균주들과 콘샐러드 분리균주들이 서로 다른 2개의 소그룹으로 나뉘었다. REP-PCR 결과 또한 2개 그룹 사이에 차이를 보여주었다. 기주특이성, 기질이용성 그리고 일부 유전적 특성들은 A. valerianellae 내에 병원성이 다른 2가지 그룹 즉 수박분리균주 그룹과 콘샐러드 분리균주 그룹이 존재함을 보여주고 있다.
Conflicts of Interest
No potential conflict of interest relevant to this article was reported.