국내의 토마토 주요 바이러스 진단을 위한 역전사중합반응법용 프라이머 세트
Specific Primer Sets for RT-PCR Detection of Major RNA Viruses of Tomato Plants in Korea
Article information
Abstract
Major tomato viruses in Korea are Tomato chlorosis virus (ToCV), Tomato spotted wilt virus (TSWV), Cucumber mosaic virus (CMV), Pepper mottle virus (PepMoV), and Tomato mosaic virus (ToMV). RT-PCR conditions for the viruses were examined, especially in primer set and RT-PCR mixture. Total 46 primer sets from the unique sequence of the viruses were tested for nonspecific background products in a RT-PCR mixture without template. Among them 16 primer sets were applied to healthy tomato RNA, resulting the compatibility between RT-PCR mixture and primer set influenced RT-PCR to reduce nonspecific background products. Based on the combinations among cDNA synthesis parameters and RT-PCR mixtures, two reaction mixtures were finally selected for ToCV detection. The condition allowed to determine more specific primer sets; C029 (ToCV), C072 (TSWV), C070 (CMV), C048 (PepMoV), and C065 (ToMV). These primer sets are expected to be of use to specific detection of the major viruses in tomato plants.
서론
국내의 토마토에서 발생하는 주요 바이러스는 Tomato chlorosis virus (ToCV), Tomato spotted wilt virus (TSWV), Cucumber mosaic virus (CMV), Pepper mottle virus (PepMoV), Tomato mosaic virus (ToMV), Tomato yellow leaf curl virus (TYLCV)이며, TYLCV를 제외한 나머지는 모두 RNA 바이러스이다(Ji 등, 2008; Kataya 등, 2008; Kim 등, 2011). ToCV는 2013년에 논산, 익산, 함평, 화순, 제주에서 처음 발생하였고, 감염된 식물체는 잎갈변과 엽맥퇴록 병징을 보이며, 7종 이상의 잡초에서 감염이 확인되었다(Kil 등, 2015). TSWV는 2004년 안양 관양동 지역에서 대발생하였고, 감염식물에 괴사 및 다중 원형반점을 일으킨다(Cho 등, 2005). CMV는 국내 채소류에서 가장 많이 발생하는 바이러스로 토마토에서는 잎에 모자이크와 고사리잎(fern leaf) 모양 병징을 일으킨다(Kim 등, 2011). 이밖에 PepMoV와 ToMV는 토마토 품종에 따라 황화 모자이크, 엽맥괴사, 퇴록반점 등을 일으키는 것으로 알려져 있다(Kim 등, 2008).
식물바이러스 진단은 바이러스 피해를 최소화하기 위한 초기 대응책이며(Aboul-Ata 등, 2011), 효소면역항체법(enzyme-linked immunosorbent assay, ELISA)에서부터 차세대염기서열분석(next generation sequencing, NGS)법까지 다양한 방법이 이용된다(Boonham 등, 2014; Jeong 등, 2014). 특히, 역전사중합반응(RT-PCR)법은 혈청학적 진단법에 비해 검출감도와 특이도가 높다(Hu 등, 1995; Mumford 등, 1994; Park과 Kim, 2004; Webster 등, 2004). 또한 실험절차가 간편하고 진단에 필요한 프라이머 설계 및 제작이 용이하여 식물바이러스 진단에 널리 이용된다(Webster 등, 2004). 일반적으로 식물바이러스 진단용 프라이머는 대상 바이러스 자체 또는 그와 유사한 계통의 유전체서열을 참고하여 설계한다. RT-PCR 진단에서 기주의 유전체서열을 고려하지 않고 설계된 프라이머를 사용할 경우, 프라이머 염기서열과 상동성이 있는 기주유래 비특이 산물의 생성을 완전히 회피하는 것은 매우 어려운 일이다. 따라서 RT-PCR의 진단 효율을 높이기 위해서는 목적 바이러스에 특이적인 프라이머 선발, 주형 준비법과 사용량, cDNA 합성온도와 시간, RT-PCR 반응액의 종류, 프라이머 및 PCR 증폭 조건 등을 고려해야 한다(Chen 등, 2011; Hassani-Mehraban 등, 2016; Kumar 등, 2011; Lee 등, 2011; Okuda와 Hanada, 2001; Webster 등, 2004). 이들 가운데 프라이머는 진단의 특이성에 가장 많은 영향을 주는 요인이다(Hassani-Mehraban 등, 2016; Kumar 등, 2011; Mumford 등, 1994; Webster 등, 2004). 오늘날에는 NGS 기술 발달로 과거 10여 년 전에 비해 식물바이러스뿐만 아니라 다양한 작물의 유전체 정보가 풍성해졌다(Thottathil 등, 2016). 2012년에는 국내 과학자가 포함된 국제협력연구를 통해 토마토의 전장유전체서열이 완전히 분석되었다(The Tomato Genome Consortium, 2012). 이는 기주의 전장유전체 정보를 고려해 토마토에 발생하는 바이러스에 특이적인 프라이머 설계가 가능함을 의미한다. 따라서 본 연구에서는 토마토 전장유전체서열, expressed sequence tag (EST), 식물바이러스 염기서열 간 상동성 분석을 통해 바이러스 특이적인 프라이머 후보군을 탐색하고, one step RT-PCR mix와 cDNA 합성 조건을 달리하여 토마토 주요바이러스 진단에 필요한 특이 프라이머 세트를 선발하고자 하였다.
재료 및 방법
식물 및 바이러스
바이러스 증식 및 건전 RNA 채취를 위해 국내에 시판되는 토마토 두 품종(서광과 주옥)을 사용하였다. 바이러스는 농촌진흥청 작물보호과 식물바이러스실에서 유지하고 있는 ToCV-tom, PepMoV-tom, ToMV-n1을 사용하였고, TSWV-old와 CMV-P1은 농우바이오(Nongwoo Bio, Suwon, Korea) 이장하 박사로부터 분양 받았다.
바이러스 증식 및 주형 준비
감염 RNA 채취를 위해 각각의 바이러스가 감염된 토마토 또는 담배 잎을 접종원으로 사용하여 TSWV는 주옥 토마토에, 나머지 바이러스는 서광 토마토에 증식하였다. 접종액은 멸균한 막자사발에 감염잎(1 g)과 접종액(0.1 M Tris-HCl, pH 8.0, 1% sodium sulfite) 3 ml를 널고 마쇄한 다음, 일반 두루마리 화장지 4겹으로 여과하여 준비하였다. 접종개체는 적정량의 carborundum (600 mesh)을 혼합한 여과액을 묻힌 붓으로 떡잎 또는 본엽 1-2매 전개된 잎을 수회 문질러 준비하였고, 흐르는 물로 곧바로 세척한 다음 과천 소재 실험 농장에서 재배하였다. 접종 후 25일경 접종엽을 제외한 나머지 잎을 일괄 채취하고, 필요한 양 만큼 알루미늄 호일로 포장한 다음 -20°C에 보관하면서 필요 시 꺼내어 사용하였다. 식물체 전체 RNA는 제품 사용설명서에 따라 BCS Plant RNA Prep Kit (BioCube System, Gwacheon, Korea)로 정제하였고, 정제한 RNA는 -20°C에 보관하면서 분리 후 7일 이내에 사용하였다. RNA 농도는 Nanodrop ND2000 (Thermo Scientific, Waltham, MA, USA)를 이용하여 100-600 ng/µl 농도가 되도록 필요한 농도로 조절하여 사용하였다.
검정용 프라이머 분석 및 제작
National Center for Biotechnology Information (NCBI)에 등록된 바이러스 염기서열을 분석한 다음 전장 염기서열이 보고된 대표 바이러스를 선발하고(Table 1), 생물정보분석 회사인 씨더스(Seeders, Daejeon, Korea)에 분석 의뢰하였다. 분석방법을 간단히 요약하면, 바이러스 유전체서열과 토마토 전장유전체서열, 토마토 EST, 2014년 11월까지 NCBI에 등록된 모든 식물바이러스 유전체 염기서열(plant virus genome sequence data, PVS data) 간의 상동성 분석을 통해 5가지 바이러스 특이 서열을 1차로 선발하였다. 프라이머는 선발 서열과 Primer3 프로그램(Koressaar와 Remm, 2007)으로 예상 증폭산물의 크기가 500-1,500 bp, Tm값은 60°C, 나머지 조건은 프로그램의 기본값에 적합하도록 설계한 다음, 마크로젠사(Macrogen, Seoul, Korea)에 의뢰하여 합성하였다.
RT-PCR 반응액과 증폭산물 확인
RT-PCR 반응액은 Taq DNA polymerase의 농도(1 U/10 µl 또는 2 U/10 µl)와 활성 억제 방식을 달리하여 cDNA 합성과 DNA 증폭이 동일 반응액에서 일어나도록 one-step 방식으로 제조하였다. Taq DNA polymerase 활성 억제 방식은 RT-PCR mix1과 2는 chemical inhibition 방식, RT-PCR mix3과 4는 antibody inhibition 방식이며, 반응액에 포함된 Taq DNA polymerase의 농도는 RT-PCR mix1과 3은 1 U/10 µl, RT-PCR mix2와 4는 2 U/10 µl가 되도록 제작하였다.
cDNA 합성 및 DNA 증폭은 PCR기(Life Eco; Bioer Technology, Hangzhou, China)에서 수행하였다. 증폭산물 가운데 10 µl를 EtBr 혹은 GelGreen (BioCube System)이 첨가된 1% agarose gel에서 100 V 5 A 조건으로 30-60분간 전기영동하고, 반응 결과는 gel-documentation system GDS200D (iNtRON, Seongnam, Korea)로 확인하였다.
RT-PCR mix와 프라이머 조합별 특이도 비교
첫 번째 실험에서는 식물 RNA 연구용으로 시판되고 있는 RT-PCR mix3과 공시 프라이머 조합 간 반응양상을 조사하였다. RT-PCR 반응액은 주형을 첨가하지 않고, 2× RT-PCR mix 10 µl, 멸균수 8 µl, 10 pmol/µl 농도의 정방향과 역방향 프라이머를 각각 1 µl씩 첨가하여 제조하였다. 반응은 42°C/20분, 95°C/10분, 35회(95°C/1분, 56.0°C, 59.6°C, 63.7°C 처리구에서 1분, 72°C/1분)로 수행하였으며, 반응양상은 비특이 증폭산물 유무와 정도(약함과 강함)에 따라 판별하였다.
두 번째 실험에서는 첫 번째 실험에서 선발한 프라이머 조합을 대상으로 특이성을 심층 조사하였다. 건전한 서광 토마토의 RNA (100 ng/µl) 1 µl를 주형으로 사용하였고, RT-PCR mix 종류와 Tm값 구배 조건(51.0°C, 52.2°C, 53.9°C, 56.0°C, 58.5°C, 60.7°C, 62.4°C), 멸균수 7 µl 첨가를 제외한 나머지 실험 조건은 첫 번째 실험과 동일하게 하였다. 비특이 정도는 비특이 증폭산물의 총 개수, 밴드 진하기, dimer의 진하기와 넓이 등을 고려하여 육안으로 판별하였다. 선발한 프라이머 개수가 적었던 ToCV와 TSWV 경우는 조합별 결과를 젤 그림상에서 비교하였고, PepMoV, ToMV, CMV 프라이머 조합은 다음과 같은 조건으로 비교하였다. 프라이어별로 세 가지 Tm값(56.0°C, 59.6°C, 63.7°C)에서 발생한 비특이 증폭산물의 총 개수(A), 1 kb DNA ladder의 500 bp 밴드의 진하기를 1로 했을 때 증폭산물의 상대적인 진하기(B), 1 kb DNA ladder의 100-200 bp 사이의 구간 넓이를 3으로 했을 때 dimer의 상대적인 진하기와 넓이(0: 없음, 1: 얇거나 흐림, 5: 넓거나 아주 진함) (C)를 A×+C 형식으로 수치화하였다. 그리고 이들 값을 바이러스별로 묶어 평균값으로 환산하고 오차 값을 계산한 다음 그 값이 작을수록 특이도가 높은 것으로 판독하였다.
RT-PCR mix 종류와 주형농도, cDNA 합성온도 및 시간별 반응양상 비교
본 실험에서는 4가지 RT-PCR mix와 ToCV 진단용 프라이머 조합 가운데 주형을 넣지 않은 상태에서 가장 특이도가 높았던 C029 프라이머 조합을 사용하였다. 특이성은 이전처럼 비특이 증폭산물을 A×+C 형식으로 수치화한 다음 판별하였다. 주형 농도, cDNA 합성온도와 시간 변화에 따른 특이도 비교는 다음의 조건 1, 2, 3에 따라 각각 수행하였다.
조건 1: RT-PCR 반응액은 2× RT-PCR mix 10 µl, 멸균수 7 µl, 10 pmol/µl 농도의 정방향과 역방향 프라이머를 각각 1 µl, 농도를 달리한 RNA (100, 200, 400, 800 ng/µl) 1 µl를 첨가하여 제조하였다. cDNA 합성은 42°C/20분, PCR 반응은 95°C/10분, 35회(95°C/1분, 58°C/1분, 72°C/1분)로 수행하였다.
조건 2: RT-PCR 반응액은 조건 1의 주형 농도를 100 ng/µl로 고정하여 제조하였고, cDNA는 42°C, 45°C, 48°C, 51°C, 56°C 온도 처리구에서 20분간 합성하였으며, PCR 반응은 조건 1과 동일하게 수행하였다.
조건 3: cDNA는 48°C에서 각각 5, 10, 15, 20분간 합성하였고, 나머지는 조건 2와 동일하게 실험하였다.
선발 프라이머 검출감도 및 특이도 비교
건전 RNA와 감염 RNA는 토마토와 주옥(TSWV의 경우) 토마토 잎에서 분리하였는데, 원액과 이를 diethylpyrocarbonate (DEPC)-water로 10배씩 연속 희석한 용액을 주형으로 사용하였다. RT-PCR 반응액은 2× RT-PCR mix3 10 µl, 멸균수 7 µl, 10 pmol/µl 농도의 정방향과 역방향 프라이머를 각각 1 µl, RNA (약 200 ng/µl) 1 µl를 첨가하여 제조하였으며, cDNA 합성은 48°C/5분, PCR 반응은 95°C/10분, 35회(95°C/1분, 58°C/1분, 72°C/1분), 72°C/1분 1회로 수행하였다.
결과
프라이머 조합별 특이도 비교
시판 중인 RT-PCR mix3에 주형을 넣지 않고, 세 가지 Tm값(56.0°C, 59.6°C, 63.7°C)에서 46개 프라이머 조합(Table 2)의 증폭 양상을 조사하였다. C041 (ToCV), C044 (ToCV), C073 (TSWV) 프라이머 조합에서는 Tm값 변화에 상관없이 비특이 산물이 증폭되지 않았다. 반면에 ToCV의 C029, C032, C040, PepMoV의 C048, C054, C055, ToMV의 C065, C066, CMV의 C068, C069, C070, C071, TSWV의 C072 조합에서는 비특이 산물이 약하게 증폭되었고, 나머지 프라이머 조합에서는 결과 판독에 영향을 줄 정도로 비특이 산물이 많이 증폭되었다(Table 2).
한편, 프라이머 이외에 다른 반응 조건을 최적화한다면 비특이 산물이 조금 생성된다 할지라도 목적바이러스 진단에 사용할 수 있을 것으로 판단되었다. 따라서 비특이 산물이 증폭되지 않거나 약하게 증폭된 프라이머 조합을 이후 실험재료로 선발하였다.
진단용 RT-PCR mix 선발
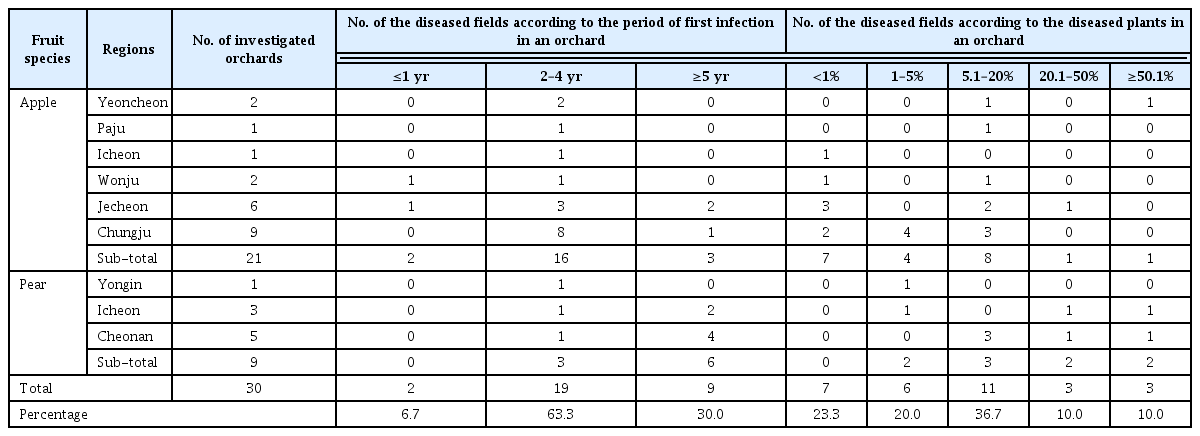
이전 실험에서 선발한 프라이머 조합과 건전한 서광 토마토 RNA를 이용하여 RT-PCR mix별 비특이 산물 생성 정도를 조사하였다. ToCV 진단용 프라이머의 경우 RT-PCR mix별 증폭양상은 비슷하였다. C041의 경우 주형을 넣지 않은 상태에서는 비특이 산물이 증폭되지 않았으나(Table 2), 건전한 RNA 주형이 존재할 때는 특이도가 다소 떨어졌다(Fig. 1). TSWV 진단용 프라이머 조합은 RT-PCR mix 간에 비특이 산물 생성 정도가 다르게 나타났는데, C072는 RT-PCR mix1과 4에서 목적산물 범위에 비특이 산물이 많이 생성되었고, C073은 RT-PCR mix4에서 비특이 정도가 가장 심하였다(Fig. 1). 대체적으로 ToCV와 TSWV 프라이머 조합에서는 RT-PCR mix2와 3이 RT-PCR mix1과 4에 비해 다소 양호한 결과를 보였으나, C072의 경우 RT-PCR mix2와 3 간의 특이도 정도를 gel 그림으로 세부 비교하는 것에는 다소 어려움이 있었다(Fig. 1).
Gel images of nonspecific background products on four different RT-PCR mixtures. RT-PCR was performed using 1 μl of RNA (100 ng/μl) from healthy tomato of ‘Seogwang’ as template. The condition was as follows; cDNA synthesis (42°C/20 min), denaturation (95°C/10 min), amplification (35 cycles of 95°C/1 min, 51.0°C–62.4°C/1 min, and 72°C/1 min), and final extension (72°C/10 min). Each gel is divided into eight lanes; lane M for 100 bp DNA ladder, lane 1–7 for 51.0°C, 52.2°C, 53.9°C, 56.0°C, 58.5°C, 60.7°C, and 62.4°C in order. The label at left side of each gel indicates target virus name and primer combination/expected size of amplicon. ToCV, Tomato chlorosis virus; TSWV, Tomato spotted wilt virus.
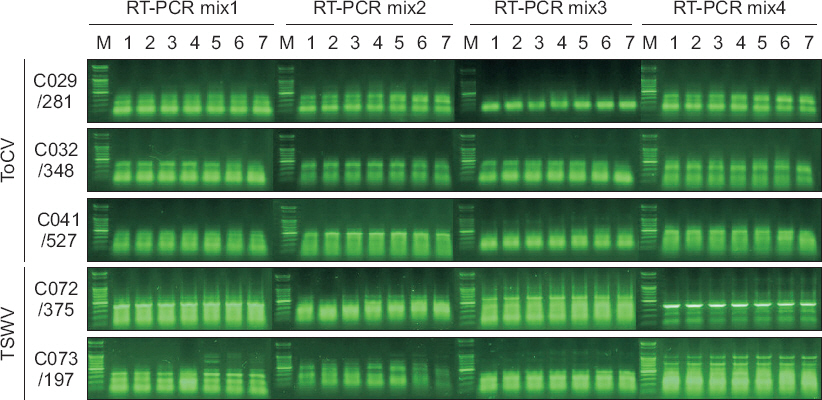
따라서 PepMoV, ToMV, CMV 프라이머 조합에서는 재료 및 방법에 서술한 것처럼 프라이머 조합의 비특이 정도를 바이러스별 평균값과 오차값의 크기로 비교하였다. PepMoV와 CMV 프라이머 조합의 경우 ToCV 및 TSWV용 프라이머 조합에서처럼 대체적으로 RT-PCR mix3에서 양호한 결과를 보였으며, ToMV용 프라이머 조합에서는 RT-PCR mix1이 가장 특이도가 높은 것으로 나타났다(Fig. 2). 한편, Taq DNA polymerase 활성 억제 방식에 따른 비특이 정도를 비교했을 때 chemical inhibition 방식의 반응액(RT-PCR mix1과 2)이 antibody inhibition 방식(RT-PCR mix3과 4)보다 다소 높게 나타났으며, 동일방식의 반응액 간에는 Taq DNA polymerase의 농도가 1 U/10 µl일 때 2 U/10 µl보다 비특이 정도가 낮게 나타났다(Fig. 2).
Bar graph for average value of non-specificity in different RT-PCR mixtures. The average value was calculated from amount and intensity of nonspecific background products in each primer combinations for target virus. RT-PCR was performed at the condition described in Fig. 1 except for Tm value (56.0°C, 59.6°C, and 63.7°C). (A) PepMoV, Pepper mottle virus. (B) ToMV, Tomato mosaic virus. (C) CMV, Cucumber mosaic virus.
cDNA 합성 조건 최적화 및 선발 프라이머 적용
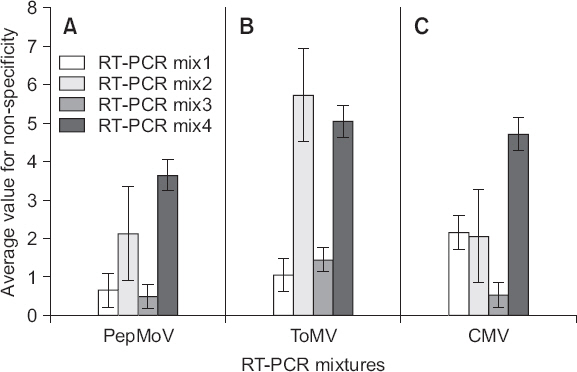
RT-PCR을 이용한 진단에서 cDNA 합성은 진단의 특이성을 결정짓는 중요한 단계이다. cDNA 합성에 적합한 조건을 찾고자 공시한 4종의 RT-PCR mix에서 주형의 농도, cDNA 합성온도 및 반응시간 변화에 따른 비특이 정도를 비교하였다. 이를 위해 주형을 넣지 않은 RT-PCR mix3에서 비특이 정도가 낮고, 건전한 RNA 주형 첨가 시 비특이 산물이 약하게 생성된 ToCV용 C041이 적합하다고 판단되어 본 실험에 사용하였다. 각각의 RT-PCR 반응액에서 주형(서광 토마토 건전주의 RNA)을 농도별로 첨가하고 비특이 정도를 조사한 결과, 비특이 정도는 RT-PCR mix1, 3, 2, 4 순으로 낮게 나타났으며, RT-PCR mix4를 제외한 나머지 반응액에서는 주형의 농도 변화에 관계없이 일정하였다. 특이한 것은 RT-PCR mix4의 경우 주형의 농도가 200 ng/µl 이하일 때보다 400 ng/µl 이상 처리구에서 비특이 정도가 더 높게 나타났다(Fig. 3A).
Bar graph for average value of non-specificity according to the combinations between cDNA synthesis parameter and reaction mixture in RT-PCR. The average value was calculated from amount and intensity of nonspecific background products in each primer combination. RT-PCR was performed with C029 primer combination of Tomato chlorosis virus at the condition described in Fig. 1 with exceptions; cDNA synthesis (48°C/20 min) (A), template RNA (100 ng/μl) from healthy tomato of ‘Seogwang’ (B), and cDNA synthesis (20 min) (C). Tm value for PCR is 58°C over all test.
주형의 농도와 cDNA 합성시간을 100 ng/µl와 20분으로 고정하고 온도변화에 따른 비특이 정도를 조사했을 때, RT-PCR mix4를 제외한 나머지 반응액에서는 권장 온도인 48°C 이하에서 비슷한 경향을 보였으나, 역전사효소의 활성이 떨어지는 51°C 이상에서는 RT-PCR mix3이 다른 반응액에 비해 영향을 많이 받았다(Fig. 3B).
한편, cDNA 합성시간에 따른 비특이 정도는 RT-PCR mix1을 제외한 나머지 반응액에서 합성시간이 길어질수록 증가하는 경향을 보였다. 비특이 정도는 RT-PCR mix1이 전 시간대에서 가장 낮았고, 합성시간 10분에서 RT-PCR mix3은 RT-PCR mix1과 비슷한 결과를 보였다(Fig. 3C).
RT-PCR mix1의 경우 RT-PCR mix3에 비해 기능이 다소 우수하였으나, 최적화된 RT-PCR 조건에서는 큰 차이가 없을 것으로 판단되어 RT-PCR mix3을 이용해 다음 실험을 진행하였다. RT-PCR mix3에 적합한 조건을 이전 실험에서 비특이 산물이 생성되지 않거나(C041, C044, C073) 그 정도가 약했던(C029, C032, C040, C048, C054, C055, C065, C066, C069, C070, C071, C072) 프라이머 조합에 적용했을 때, 바이러스별로 ToCV는 C029, TSWV는 C072, CMV는 C070, PepMoV는 C048, ToMV C065의 특이도와 민감도가 가장 좋았다(data not shown). 이를 기준으로 바이러스 진단을 위해 선발한 프라이머 조합의 전체적인 결과를 보면 C029 (ToCV), C048 (PepMoV), C070 (CMV)은 건전과 감염 RNA 원액과 희석액 모두에서 비특이 산물이 전혀 생성되지 않았고, 결과에 영향을 줄 정도는 아니었으나 C072 (TSWV)와 C065 (ToMV)는 고농도 시료에서 비특이 산물이 증폭되었다(Fig. 4).
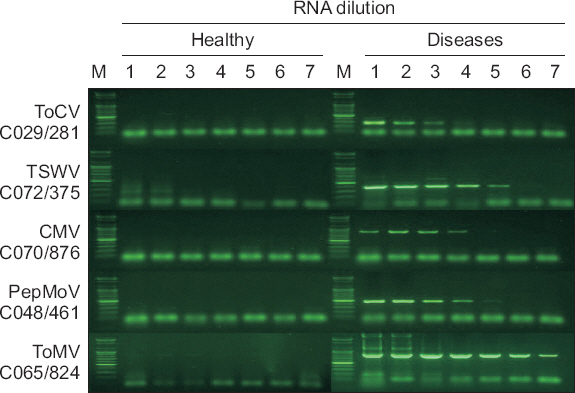
RT-PCR detection of five tomato viruses using finally selected primer combinations for each virus. Each gel is divided into eight lanes; lane M for 100 bp DNA ladder, lane 1 for initial sap of purified total RNA, and lanes of 2–7 for 10-fold serial dilutions. The label at left side of each gel indicates target virus name and primer combination/expected size of amplicon. Template RNA was prepared from ‘Seogwang’ tomato plant except for TSWV (‘Juok’ tomato). RT-PCR was performed as follows: cDNA synthesis (47°C/5 min), denaturation (95°C/10 min), amplification (35 cycles of 95°C/1 min, 65°C/1 min, and 72°C/1 min), and final extension (72°C/10 min). ToCV, Tomato chlorosis virus; TSWV, Tomato spotted wilt virus; CMV, Cucumber mosaic virus; PepMoV, Pepper mottle virus; ToMV, Tomato mosaic virus.
고찰
본 연구에서는 바이러스 특이 프라이머 조합을 선발하고자 토마토와 대상 바이러스의 염기서열 상동성을 기초로 바이러스 특이 염기서열을 선발하고 프라이머를 제작하였다. 그럼에도 불구하고 주형을 넣지 않은 RT-PCR 반응에서 프라이머 조합에 따라 비특이 산물이 생성됨을 확인하였다(Table 2). 일반적으로 PCR mix에는 Taq DNA polymerase 정제 방법에 따라 발현용 벡터 DNA 잔존물이 남게 되는데(Chen 등, 2015), 반응액 내에 존재하는 벡터 DNA 잔존물과 건전 RNA 간의 간섭현상 등이 비특이 산물 생성의 주요 원인 가운데 하나일 것으로 판단되었다.
다만 본 실험 결과는 비특이 산물이 많이 생성될 수 있도록 RT-PCR 검정 조건이 최적화되지 않은 상태에서 수행하였다. 그러므로 C072처럼 주형이 없거나 건전한 RNA 첨가 조건에서 일부 비특이 산물이 생성된다고 해서 진단에 활용할 수 없는 것은 아니다(Fig. 3). 진단 대상이 되는 바이러스에 보다 특이적인 프라이머를 선발하기 위해서는 기주식물과 바이러스뿐만 아니라 Taq DNA polymerase 단백질 발현시스템인 대장균 유전체도 함께 분석할 필요가 있다고 판단된다.
한편, 프라이머 종류와 함께 RT-PCR mix의 조성과 종류에 따라 진단의 특이성에 차이를 보였는데, TSWV 진단용 C072는 RT-PCR mix1과 이와 조성이 다른 RT-PCR mix4에서 비특이 정도가 심하고(Fig. 1), 다른 바이러스 프라이머 조합에서는 RT-PCR mix2와 4에서 그 정도가 심하였다(Fig. 2). 이는 Taq DNA polymerase 농도가 높을 때 비특이 산물이 많이 생성된다는 이전의 보고와 관련이 있을 것으로 판단되었다(Usta 등, 2005). 반면에 ToCV 진단용 프라이머는 주형의 농도, cDNA 합성온도와 시간을 달리했을 때 RT-PCR mix 1의 특이도가 가장 높은 것으로 나타났다(Fig. 3). 이는 C072와 C029의 결과가 상충되는 것으로 RT-PCR 반응액과 선택한 프라이머 간의 친화성이 진단의 특이성에 중요한 요인임을 암시한다.
그러나 RT-PCR mix3은 cDNA 합성온도를 역전사효소의 내성 온도인 50°C 이하와 합성시간 5분 이내에서는 RT-PCR mix1과 큰 차이를 보이지 않았다(Fig. 3). 결과적으로 RT-PCR mix3은 RT-PCR mix1에 비해 적용성이 다소 높은 것으로 보인다. 끝으로 RT-PCR mix3을 이용해 선발한 프라이머의 진단 특이성을 보면, ToMV용 C065의 경우 주형이 고농도일 때 비특이 산물이 강하게 증폭되었다(Fig. 4). 이는 경우에 따라 프라이머 자체만으로 비특이 산물 생성을 억제하는 것에는 한계가 있으며, 기주 식물의 전장유전체서열 정보나 분석 방법이 아직 완전하지 않음을 의미한다고도 할 수 있다. 예로써 전장유전체 조립 시 생물정보연구자들은 대장균을 포함한 미생물 염기서열을 모두 제거한다고 하는데(personal communication), 이것이 본 실험에 어떤 영향을 주었는지는 아직 논의되어야 할 소지가 많다고 하겠다. 이런 상황에서 비특이 산물 생성을 피하기 위해서는 Taq DNA polymerase 정제 시 DNA 오염이 되지 않은 반응액을 사용하던지(Lawyer 등, 1993) 또는 단백질 프라이머 종류를 바꾸는 것도 한 방법이지만, 당장은 RNA 주형 준비 시 DNase 처리를 통한 기주 DNA 제거가 많은 도움이 될 것으로 판단된다.
요약
국내의 토마토에서 발생하는 주요 바이러스는 Tomato chlorosis virus (ToCV), Tomato spotted wilt virus (TSWV), Cucumber mosaic virus (CMV), Pepper mottle virus (PepMoV), Tomato mosaic virus (ToMV)이다. 이들 바이러스를 진단하기 위해 프라이머 세트와 반응액을 포함하는 역전사중합반응(RT-PCR)법의 조건을 조사하였다. 공시한 바이러스에 특이적인 염기서열로부터 모두 46개 프라이머 세트를 설계하고, 이를 이용해 주형을 넣지 않은 RT-PCR에서 비특이 반응을 조사하였다. 이들 가운데 16개 조합을 건전한 토마토 RNA에 적용한 결과 프라이머 세트와 RT-PCR 반응액 간의 친화성이 비특이 반응 감소에 영향을 주었다. cDNA 합성과 관련된 인자와 RT-PCR 반응액 사이의 조합을 근거로 ToCV 진단을 위한 두 종류의 반응액을 선발하였다. ToCV 진단 시 수립된 조건을 나머지 바이러스 진단에 적용했을 때, 특이성이 높은 프라이머 세트 C029 (ToCV), C072 (TSWV), C070 (CMV), C048 (PepMoV), C065 (ToMV)를 선발할 수 있었다. 이들 프라이머 세트는 공시한 바이러스를 특이적으로 진단하는 데 유용할 것으로 판단된다.
Acknowledgement
This research was supported by Agri-Bio industry Technology Development Program, Ministry of Agriculture, Food and Rural Affairs (114069-02-2-WT011).
References
Notes
Conflicts of Interest
No potential conflict of interest relevant to this article was reported.